Основы биоинформатики - Огурцов А.Н. 2013
Основания биоинформатики
Примеры сравнения данных
Поиск схожих последовательностей в базах данных
Прежде чем проводить анализ биологических последовательностей, необходимо эти последовательности отыскать в базах данных. Например, если вы определили последовательность нового гена или нашли в геноме человека ген, ответственный за какое-то заболевание, то вы, возможно, захотите узнать, нет ли таких генов у других видов. Идеальный метод — тот, который с одной стороны чувствителен (который определяет даже дальнее родство), а, с другой стороны, селективен (благодаря которому все полученные родственные связи — истинные).
Методы поиска в базах данных подразумевают компромисс между чувствительностью и селективностью. Находит ли метод все или большинство из последовательностей, которые на самом деле существуют, или же он упускает большую их часть? А также, сколько из выданных этим методом результатов являются неправильными?
Предположим, база данных содержит 1000 последовательностей глобина. Предположим, поиск в этой базе данных по глобинам выдал 900 находок, 700 из них действительно последовательности глобина, а 200 таковыми не являются. Про такой поиск можно сказать, что у него 300 ложных отрицательных (false negatives) результатов (упущенных, не обнаруженных последовательностей) и 200 ложных положительных (false positives) результатов (обнаруженные последовательности в действительности не являются искомыми). Уменьшая порог допустимости, мы получим меньше ложных отрицательных результатов, но больше ложных положительных результатов.
Часто лучше работать с низкими порогами, чтобы быть уверенным, что ничего из того, что могло бы быть важным, не утеряно; но тогда потребуется детальная проверка результатов, для того чтобы устранить ложные находки.
Мощным инструментом для поиска последовательностей в базах данных, по имеющейся у нас последовательности, является программа BLAST (Basic Linear Alignment Sequence Tool), которую можно использовать с сайта NCBI http://www.ncbi.nlm.nih.gov/ (рисунок 30).
Рисунок 30 - Веб-страница NCBI; ссылка на программу BLAST показана в нижнем правом углу рисунка
Переход по ссылке "BLAST" http://blast.ncbi.nlm.nih.gov/Blast.cgi показан на рисунке 31. На рисунке отображена та часть страницы, которая относится только к основным (Basic) подпрограммам. Она включает:
✵ nucleotide blast - поиск данной последовательности нуклеотидов в базах данных нуклеиновых кислот используя алгоритмы blastn, megablast, dmegablast (discontiguous megablast);
✵ protein blast - поиск данной аминокислотной последовательности в базах данных белков используя алгоритмы blastp, psi-blast, phi-blast;
✵ blastx - переводит изучаемую нуклеотидную последовательность в кодируемые аминокислоты, а затем сравнивает её с имеющейся базой данных аминокислотных последовательностей белков;
✵ tblastn - изучаемая аминокислотная последовательность сравнивается с транслированными последовательностями базы данных секвенированных нуклеиновых кислот;
✵ tblastx - переводит изучаемую нуклеотидную последовательность в аминокислотную, а затем сравнивает её с транслированными последовательностями базы данных секвенированных нуклеиновых кислот.
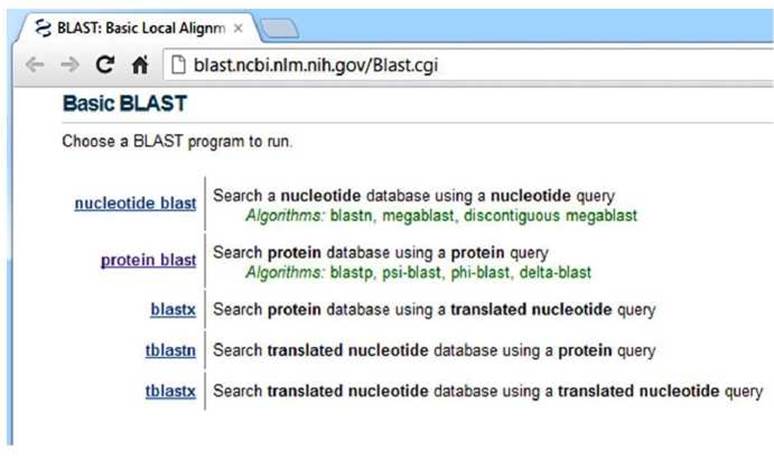
Рисунок 31 - Веб-страница программы BLAST
Здесь:
megablast - быстрое сравнение с целью поиска высоко сходных последовательностей;
dmegablast - быстрое сравнение с целью поиска дивергировавших последовательностей, обладающих незначительным сходством;
blastn - медленное сравнение с целью поиска всех сходных нуклеотидных последовательностей;
blastp - медленное сравнение с целью поиска всех сходных белковых (protein) последовательностей;
psi-blast - Position-Specific Iterated BLAST - сравнение с целью поиска последовательностей, обладающих незначительным сходством;
phi-blast - Pattern Нit Initiated BLAST - поиск белков, содержащих определённый пользователем паттерн.
Паттерн - (от англ. pattern - образец, шаблон, модель) - это либо фрагмент последовательности, либо (реже) некий стандартный набор процедур, применяемый к разным объектам.
Пример 4. Г омологи РАХ-6 гена человека.
Гены РАХ-6 контролируют развитие глаза в широком наборе видов.
Глаза человека, мухи и осьминога сильно различаются по строению. Ранее, принимая во внимание то конкурентное преимущество, которое даёт зрение, считалось, что глаза возникли независимо в каждой эволюционной ветви. Поэтому большим сюрпризом стал тот факт, что ген, контролирующий развитие человеческого глаза, имеет гомолога, управляющего развитием глаза дрозофилы.
Ген РАХ-6 был клонирован вначале у мыши и человека. Он является главным регуляторным геном, контролирующим сложный каскад событий в развитии глаза.
Мутации в гене человека вызывают клиническое состояние - аниридию - дефект в развитии глаза, при котором радужная оболочка отсутствует или деформирована.
Гомолог гена РАХ-6 в дрозофиле называется - eyeless-ген (имеет сходную функцию контроля развития глаза). Мухи, мутантные по этому гену, развиваются без глаз; и обратно, экспрессия этого гена на лапке мухи или на антенне мухи - вызывает появление эктопических (то есть находящихся не на месте) глаз. Дрозофила, мутантная по гену eyeless, была впервые описана в 1915 г. Никто и не подозревал о его родстве с геном млекопитающих. Гены насекомого и млекопитающего схожи не только по последовательности, они так близкородственны, что их активность выходит за рамки видов. Экспрессия мышиного РАХ-6 в мухе вызывает эктопическое развитие глаза, также как и собственный eyeless ген мухи.
Гомологи РАХ-6 представлены и в других классах, включая плоских червей, асцидий, морских ежей и нематод. Наблюдение, что родопсины (семейство белков, содержащих ретин в качестве хромофора) функционируют, как светочувствительные пигменты в различных классах организмов, является дополнительным доказательством общего происхождения различных систем фоторецепторов.
Настоящие структурные различия в макроскопическом строении различных глаз отражают дивергенцию и независимость развития высокоорганизованных структур.
Ген РАХ-6 человека кодирует белок, имеющий Swiss-Prot-идентификатор - Р26367. Значение этого идентификатора можно получить, если в окно поиска программы UniProt (рисунок 23) ввести "РАХ-6" и нажать "Search".
Из окна программы "BLAST" http://blast.ncbi.nlm.nih.gov/Blast.cgi (рисунок 31) запускаем "protein blast" и вводим идентификатор sp|P26367 в окно "Enter Query Sequence" (рисунок 32).
Выбираем алгоритм "PSI-BLAST" в окне "Program Selection" и запускаем поиск, нажав кнопку "BLAST" в нижнем левом углу окна (рисунок 32).
Результат поиска представляет собой огромное (в длину) окно, большую часть которого занимает список записей схожих с последовательностью, заданной для поиска, сортированный в порядке убывания статистической значимости.
Начало этого списка показано на рисунке 33.
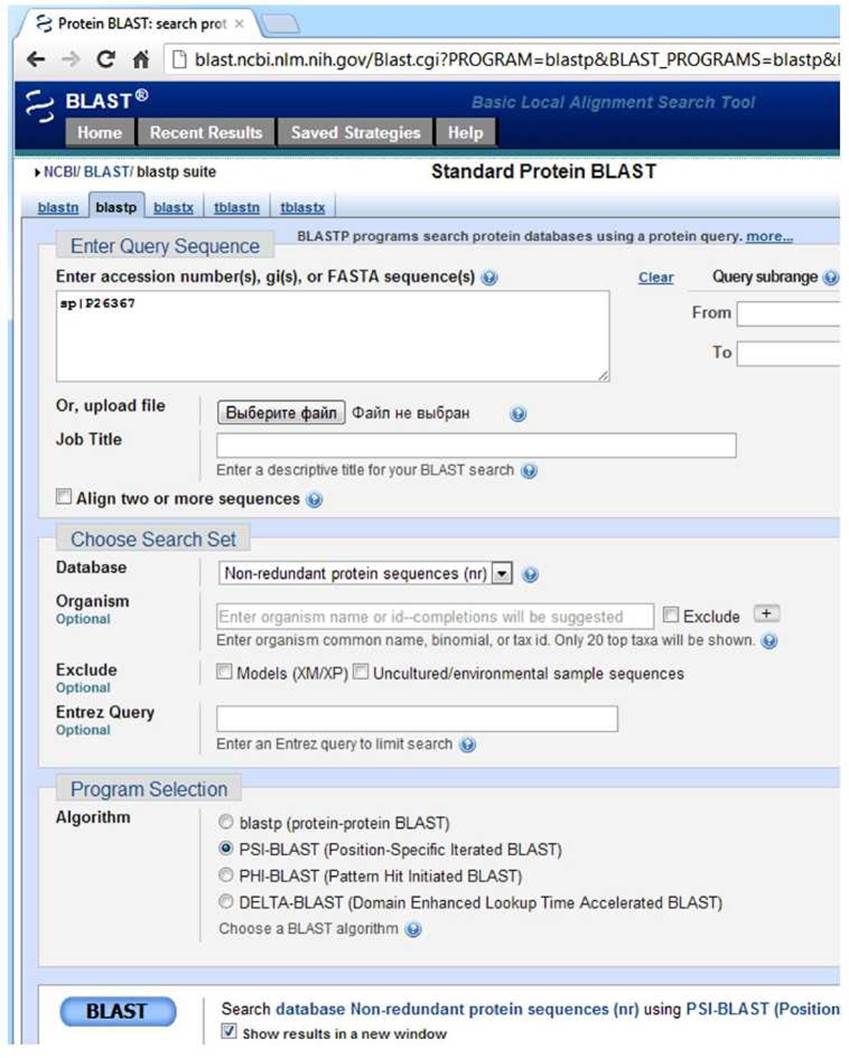
Рисунок 32 - Окно ввода программы BLAST
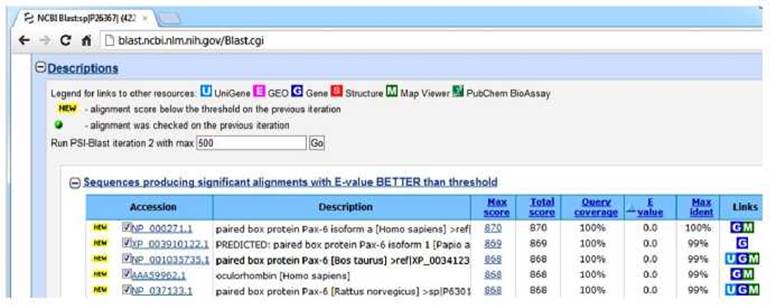
Рисунок 33 - Окно вывода программы BLAST
Каждая строка содержит одно совпадение с каким-либо геном. Рассмотрим, например, третью от начала строку, в которой приведены результаты для:

В первом столбце "Accession" располагается идентификатор гена (NP_001035735.1). Это гомолог Paired box protein Pax-6 [Bos taurus]. Базы данных ![]() обозначены в последнем столбце, в данном случае это базы данных UniGene, Gene Structure и Map Viewer.
обозначены в последнем столбце, в данном случае это базы данных UniGene, Gene Structure и Map Viewer.
Число 868 - это количество очков, присвоенное обнаруженному совпадению.
Значимость данного совпадения (E-value) измерена как Е = 0.0.
E-value (expectation value) определяется вероятностью того, что данная степень сходства может быть случайной.
E-value — это ожидаемое количество последовательностей, которые совпадут также или лучше чем данная, если поиск будет производиться базе данных такого же размера, но со случайными последовательностями.
Е = 0.0 означает полное соответствие.
Подробнее параметры выравнивания будут рассмотрены в п. 8.5.
Результат попарного выравнивания генов РАХ-6 человека и РАХ-6 буйвола (который находится в этом же окне внизу после списка совпадений) демонстрирует их абсолютное подобие (рисунок 34).

Рисунок 34 - Парное выравнивание генов РАХ-6 человека и буйвола в окне вывода программы BLAST
Чем больше значение Е, тем больше отклонений при сравнении последовательностей. Так, например, для гомолога twin eyeless Drosophila (NP_524638.3) значение E = 7∙10-145 (рисунок 35(a)).

Рисунок 35 - Схожие гены РАХ-6 человека и twin eyeless дрозофилы: а - результат поиска; б - парное выравнивание в окне вывода программы BLAST
Парное выравнивание генов РАХ-6 человека и twin eyeless дрозофилы показывает уже значительные различия в последовательностях (рисунок 35(6)).
Контрольные вопросы и задания
1. Перечислите уровни иерархии биологической номенклатуры на примере человека и плодовой мушки.
2. Какие органы называются гомологичными?
3. Чем отличается дивергентная и конвергентная эволюции?
4. На какие три империи разделил все организмы Карл Вёзе, основываясь на анализе рибосомных РНК?
5. Какими символами в окне результатов программы ClustalW2 обозначаются: одинаковая аминокислота; сходные аминокислоты; вставки; отсутствие сходства в последовательностях?
6. Чем различаются подобие и гомология последовательностей?
7. Что такое ложные отрицательные и ложные положительные результаты поиска?
8. Для чего предназначена программа BLAST?
9. Что такое nucleotide blast?
10. Перечислите алгоритмы, которые используются в nucleotide blast.
11. Что такое protein blast?
12. Перечислите алгоритмы, которые используются в protein blast.
13. Что такое blastx?
14. Что такое tblastnl
15. Что такое tblastx?
16. Что такое megablast?
17. Что такое psi-blast!
18. Что такое паттерн?
19. Что такое E-value Е-значение) последовательностей?