Основы биоинформатики - Огурцов А.Н. 2013
Основания биоинформатики
Геномы и протеомы
Методы секвенирования ДНК
Известно несколько методов определения порядка нуклеотидов в ДНК. Один из таких методов называют секвенированием с обрывом цепи, или дидезокси-секвенированием, или же (в честь его изобретателя Фредерика Сангера (Frederick Sanger)) методом полимерного копирования по Сангеру (см. [7], п. 11.2) (рисунок 41).
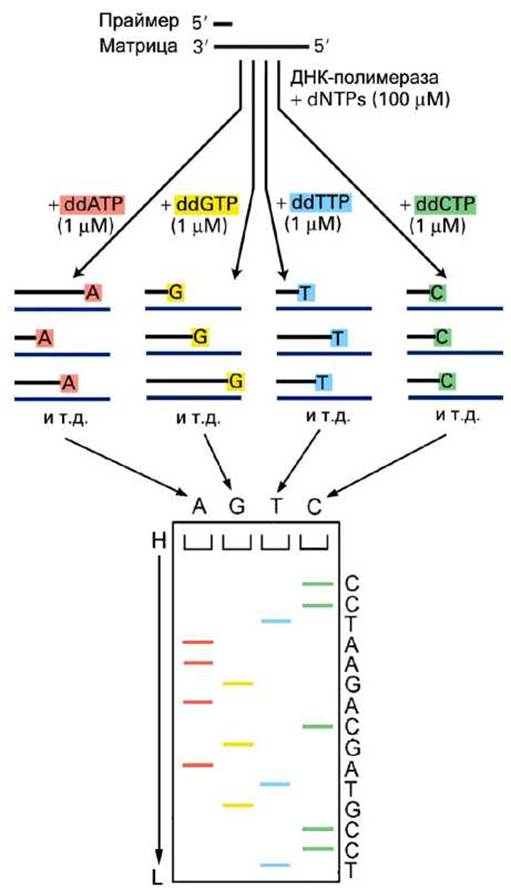
Рисунок 41 - Схема секвенирования ДНК по методу Сангера
В основной реакции секвенирования участвуют следующие реагенты: однонитевая матрица ДНК; праймер для инициации полимеризации синтезируемой цепи; четыре дезоксирибонуклеозидтрифосфата, дНТФ (dATP, dGTP, dTTP и dCTP); четыре дидезоксинуклеозидтрифосфата, ддНТФ (ddATP, ddGTP, ddTTP и ddCTP); фермент ДНК-полимераза, который встраивает комплементарные нуклеотиды в растущую нить ДНК, используя матричную нить в качестве шаблона.
Секвенирование ДНК по методу дидезокси-терминации цепи, предложенному Сангером, начинается с денатурации двойной спирали ДНК-фрагмента для того, чтобы получить одиночные матричные нити для синтеза ДНК in vitro.
Синтетический олигодезоксинкулеотид используется в качестве праймера для четырёх независимых реакций полимеризации, каждая с использованием малой концентрации одного из четырёх ддНТФ в дополнение к высокой концентрации нормальных дНТФ) (рисунок 41).
В каждой из реакций ддНТФ случайным образом присоединяется к растущей цепи ДНК в позиции соответствующего дНТФ, прекращая дальнейшую полимеризацию в данной позиции. В каждой из четырёх реакционных смесей синтезируется набор фрагментов ДНК разной длины: с общим началом и концами в определённых (одного и того же вида, но стоящих в разных позициях последовательности) основаниях. Полученную в каждой реакции смесь укороченных фрагментов денатурируют и анализируют методом гель-электрофореза.
Дидезокси-метод секвенирования ДНК полностью автоматизирован. Каждую реакционную смесь метят специфической флуоресцентной меткой (или на праймере, или на субстрате одного из нуклеотидов, например, на специфическом для данной смеси дидезоксинуклеозидтрифосфате), что впоследствии позволит определить концевые основания всех фрагментов с помощью сканера.
Затем все четыре смеси реагентов объединяют в общей ёмкости и фрагменты ДНК разделяют путём электрофореза в полиакриламидном геле (Polyacrylamide Gel Electrophoresis, PAGE), в котором меньшие фрагменты ДНК движутся быстрее, чем более крупные.
Таким методом набор фрагментов ДНК разделяется по размеру. Разрешающая способность метода PAGE позволяет разделять полинуклеотиды при разнице длин всего лишь в один остаток. Около конца дорожек сканер (фотодетектор) считывает флуоресцентную метку с проходящего мимо фрагмента ДНК, и эта информация преобразуется в данные сопоставления дорожек, представленные в виде графика, построенного из группы цветных пиков, соответствующих определённым основаниям (рисунок 42).
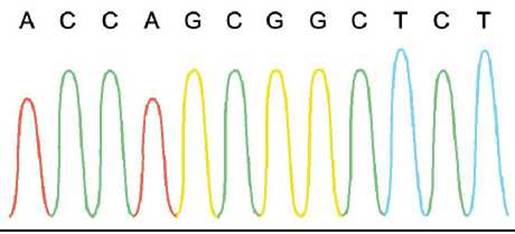
Рисунок 42 - Образец высококачественного графика сопоставления дорожек. Пики обычно распечатываются разным цветом с целью облегчения визуальной интерпретации. Программное обеспечение типа Phred считывает пики и присваивает им значения нуклеотидов А, С, G и Т
Расшифрованные последовательности ДНК хранятся в базах данных. Существуют базы данных различных ДНК-последовательностей - геномной ДНК; комплементарной кДНК; рекомбинантной ДНК. Секвенирование генома выполняют с помощью метода дробовика, метода "прогулки по хромосоме" или стратегии сборки UTR-клонов (UnTranslated Region - нетранслируемые области). Для проверки качества расшифрованных последовательностей применяют многие различные программы, например: Phred, Vector_clip, CrossMatch, RepeatMaster, Phrap и Staden-Gap4.
Появление высокопроизводительной технологии автоматизированного секвенирования ДНК с флуоресцентными метками привело к быстрому накоплению информации о последовательностях. Эта информация, в свою очередь, обеспечивает основу для получения данных о последовательностях белков вычислительными методами.
На анализе последовательности ДНК основываются множество видов исследований; например, к ним можно отнести: обнаружение филогенетических связей; генная инженерия и составление рестрикционных карт; определение структуры гена посредством предсказания нитронов и экзонов; анализ кодирующей белок последовательности с помощью открытой рамки считывания ORF (Open Reading Frame) и т. д.
Согласно основной догме молекулярной биологии ДНК транскрибируется в РНК, которая затем транслируется в белок (рисунок 43).
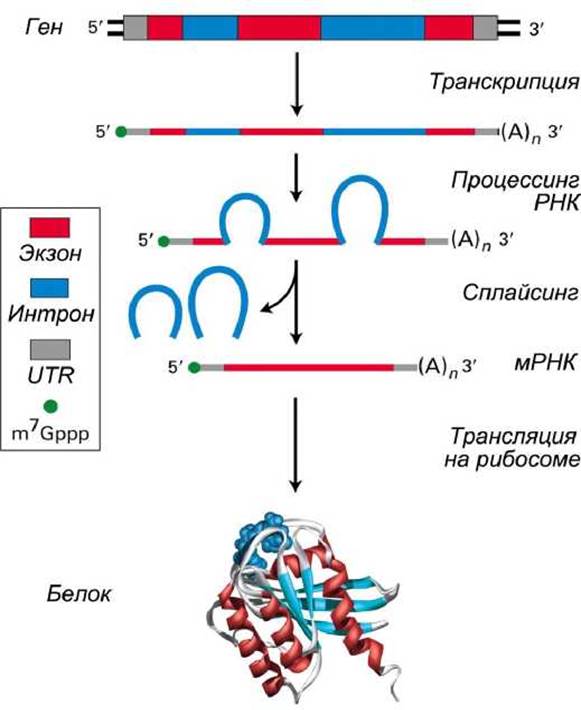
Рисунок 43 - Схема экспрессии гена
В эукариотических системах экзоны формируют часть конечной кодирующей последовательности (coding sequence), тогда как нитроны, хотя и транскрибируются, но вырезаются механизмами сплайсинга прежде, чем мРНК принимает свою окончательную, зрелую форму. Базы данных последовательностей ДНК обычно содержат информацию на уровне нетранслируемых геномных последовательностей, интронов и экзонов, мРНК, кДНК и продуктов трансляции.
Нетранслируемые области UTR встречаются как в ДНК, так и в РНК. UTR представляют собой отрезки транскрибируемой последовательности, которые с обеих сторон примыкают к кодирующей последовательности и не транслируются в белок. Нетранслируемая последовательность, особенно расположенная на 3'-конце кодирующей последовательности, весьма специфична как к самому гену, так и к биологическому виду, которому свойственно наличие этой кодирующей последовательности.