ОСНОВЫ БИОХИМИИ ЛЕНИНДЖЕРА - ТОМ 3. ПУТИ ПЕРЕДАЧИ ИНФОРМАЦИИ - 2017
ЧАСТЬ III. ПУТИ ПЕРЕДАЧИ ИНФОРМАЦИИ
Скучных ферментов не бывает.
— Артур Корнберг, «С любовью к ферментам», 1975
25. МЕТАБОЛИЗМ ДНК
В качестве хранилища генетической информации ДНК занимает центральное место среди биологических макромолекул. Нуклеотидная последовательность ДНК кодирует первичную структуру всех молекул клеточной РНК и белков и через ферменты опосредованно влияет на синтез всех других клеточных компонентов. Переход информации от ДНК к РНК и белку определяет размер, форму и жизнедеятельность всех живых существ.
ДНК прекрасно обеспечивает стабильное хранение генетической информации. Однако понятие «стабильное хранение» означает статичную неизменную картину. Оно не отражает сложности процессов, с помощью которых генетическая информация сохраняется и передается от одного поколения клеток к следующему. Метаболизм ДНК включает как процессы точного копирования молекул (репликация), так и процессы, которые влияют на саму структуру, несущую информацию (репарация и рекомбинация). Именно эти процессы мы и рассмотрим в данной главе.
Метаболизм ДНК должен происходить идеально точно. Химические реакции присоединения одного нуклеотида к другому при репликации ДНК элегантны и на удивление просты. Сложности, как мы увидим далее, в ферментативном аппарате, обеспечивающем точность передачи генетической информации. Ошибки, возникающие при синтезе ДНК, могут привести к серьезным последствиям, причем не только потому, что они изменяют или нарушают функцию определенного гена, но и потому, что такое изменение передается по наследству.
Ферменты, участвующие в синтезе ДНК. копируют молекулы ДНК, содержащие миллионы оснований. Они проделывают это с необыкновенной точностью и скоростью, несмотря на то что ДНК очень компактно упакована и связана с другими белками. Образование фосфодиэфирных связей между нуклеотидами в растущей цепи ДНК — только часть сложного процесса, в котором задействовано множество белков и ферментов.
Задача сохранения генетической информации лежит в основе процесса репарации ДНК. В гл. 8 (т. 1) подробно описана чувствительность ДНК ко многим разрушительным воздействиям. Такие воздействия происходят нечасто, но они играют очень важную роль, поскольку устойчивость организмов к изменениям последовательности ДНК очень низкая. ДНК — единственная макромолекула, для которой предусмотрена система репарации; количество, разнообразие и сложность механизмов репарации отражает многообразие пагубных для ДНК воздействий.
Клетки могут перестраивать хранящуюся в них генетическую информацию в процессах под общим названием «рекомбинация». Казалось бы, процесс рекомбинации подрывает принцип первостепенного значения стабильности и целостности генетической информации. Однако на самом деле большинство перестроек ДНК играет конструктивную роль в поддержании целостности генома, специфическим образом влияя на репликацию и репарацию ДНК и расхождение хромосом.
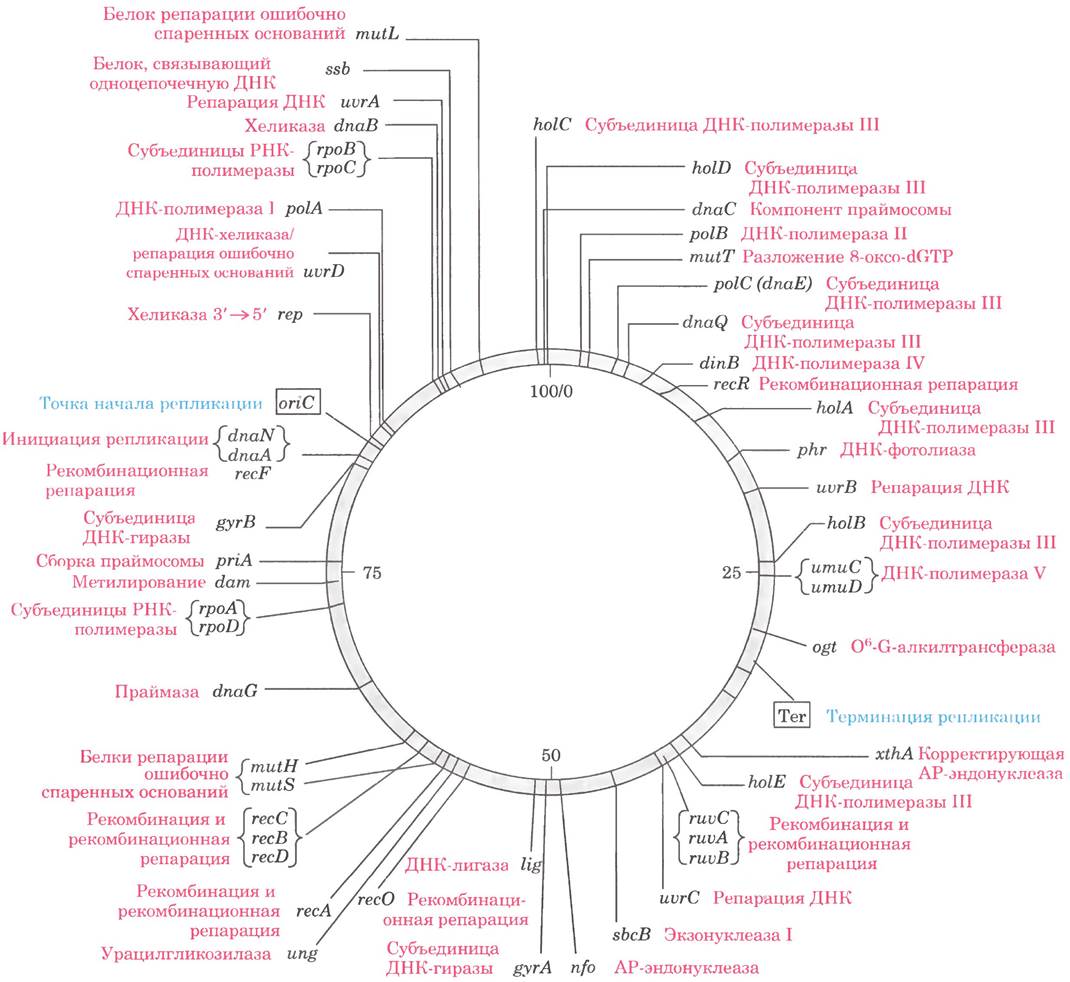
В этой главе особое внимание уделяется ферментам метаболизма ДНК. Они заслуживают внимательного изучения не только ввиду их большого биологического значения и чисто научного интереса, но и в связи с их возрастающей ролью в качестве лекарственных препаратов в медицине и в качестве инструментов в широком круге современных биохимических технологий. Многие ценные открытия в области метаболизма ДНК были сделаны на клетках Escherichia coli, поэтому для объяснения основ метаболизма обычно в качестве примера служат хорошо известные ферменты этой бактерии. Беглый взгляд на некоторые важные гены на генетической карте Е. coli (рис. 25-1) позволяет представить себе сложность ферментных систем, участвующих в метаболизме ДНК.
Рис. 25-1. Хромосомная карта Escherichia coli. Показано расположение генов, кодирующих многие важные для метаболизма ДНК белки. Количество известных генов, участвующих в метаболизме ДНК, свидетельствует о сложности этих процессов. Числа от 0 до 100 внутри кольцевой хромосомы соответствуют генетическим единицам измерения, называемым минутами. Каждая минута соответствует отрезку молекулы ДНК длиной примерно 40 000 п. н. Трехбуквеные названия генов обычно отражают некоторые аспекты их функции, к примеру, mut — мутагенез; dna — репликация ДНК; pol — ДНК-полимераза; rро — РНК-полимераза; uvr — устойчивость к УФ; rес — рекомбинация; dam — метилирование аденина; lig — ДНК-лигаза; Теr — терминация репликации; ori — точка начала репликации (у Е. coli это oriC, как на рисунке).
Прежде чем перейти к детальному изучению процесса репликации, сделаем короткое отступление и поговорим о принятых сокращениях названий бактериальных генов и белков, поскольку со многими из них нам предстоит встретиться в этой и последующих главах. Аналогичные договоренности используются и в обозначениях эукариотических генов, хотя конкретное сокращение может зависеть от вида организма, и единого правила для всех эукариотических систем не существует.
Ключевые договоренности.
Бактериальные гены обычно обозначают тремя строчными буквами курсивом, и, как правило, название отражает функцию этих генов. Например, dna, uvr и rес гены означают репликацию ДНК, устойчивость к поражающему действию УФ-облучения и рекомбинацию соответственно. Если несколько генов отвечают за одну и ту же функцию, они дополнительно обозначаются буквами А, В, С и т. д., например, dnaA, dnaB, dnaQ, что обычно указывает на порядок их открытия, а не порядок участия в цепи реакций. ■
Использование сокращений в названиях белков менее очевидно. В ходе генетических исследований обычно выделяют и характеризуют белковые продукты каждого гена. Многие бактериальные гены были идентифицированы и названы до того, как выяснилось значение их белковых продуктов. Иногда оказывается, что продукт гена — это уже известный белок, и его приходится переименовывать. Однако часто бывает, что продукт гена еще неизвестен и обладает активностью, которую нельзя описать обычным названием фермента.
Ключевые договоренности.
Бактериальные белки часто сохраняют названия соответствующих генов. Названия белков Е. coli пишутся прямым шрифтом с заглавной буквы, например, белковые продукты генов dnaA и rесА называются DnaA и RecA соответственно. ■
25.1. Репликация ДНК
Задолго до установления структуры ДНК ученых удивляла способность организмов воссоздавать самих себя и способность клеток образовывать много идентичных копий крупных и сложных макромолекул. Рассуждения на эту тему концентрировались вокруг концепции матрицы — структуры, которая позволяет молекулам соединяться в определенном порядке и образовывать макромолекулу с уникальной последовательностью и функцией. В 1940-е гг. уже сложилось представление о том, что носителем генетической информации является ДНК, но только когда Джеймс Уотсон и Фрэнсис Крик установили ее структуру, стало понятно, каким образом ДНК играет роль матрицы для репликации и передает генетическую информацию: дело в том, что одна ее цепь комплементарна другой. Основания образуют пары по определенным правилам, и каждая цепь является матрицей для новой цепи с предсказуемой комплементарной последовательностью (см. рис. 8-14, 8-15 в т. 1).
Было доказано, что основные свойства процесса репликации ДНК и каталитические механизмы этого процесса в значительной степени идентичны у всех видов организмов. Именно это единство механизмов мы постараемся подчеркнуть в нашем обсуждении, продвигаясь от общих основ процесса репликации к ферментам репликации Е. coli и, наконец, к репликации эукариот.
Основные принципы репликации ДНК
В ранних исследованиях репликации бактериальной ДНК и ее ферментов было установлено несколько базовых принципов, на которых основан синтез ДНК у всех живых существ.
Репликация ДНК полуконсервативна.
Каждая цепь ДНК служит матрицей для синтеза новой цепи, при этом образуются две новые двухцепочечные молекулы ДНК, каждая из которых состоит из одной новой и одной старой цепей. Поэтому процесс называется полуконсервативной репликацией.
Уотсон и Крик выдвинули гипотезу полуконсервативной репликации вскоре после публикации своей статьи о структуре ДНК с 1953 г.; в 1957 г. эта гипотеза была подтверждена Мэтью Мезельсоном и Франклином Сталем, которые искусно провели следующие эксперименты. Мезельсон и Сталь культивировали клетки Е. coli на протяжении многих генераций в среде, в которой единственный источник азота (NH4Cl) содержал тяжелый изотоп азота 15N вместо наиболее распространенного легкого изотопа 14N. Плотность выделенной из этих клеток ДНК примерно на 1% больше, чем у обычной [14N] ДНК (рис. 25-2, а). Несмотря на столь небольшое различие, смесь тяжелой [15N] ДНК и легкой [14N] ДНК можно разделить центрифугированием в градиенте плотности хлорида цезия.
Рис. 25-2, а — несколько поколений клеток выращивали в среде, содержащей только тяжелый азот 15N, поэтому в ДНК этих клеток содержался исключительно азот 15N, что показано в виде одной (синей) полосы, образующейся при центрифугировании в градиенте плотности CsCl. б — клетки переносили на среду, содержащую только легкий азот 14N, и после первого деления клеток выделяли ДНК, которая в градиенте хлорида цезия образовывала более высокую (фиолетовую) полосу, в — после второго цикла репликации ДНК разделялась на две полосы: гибридную (фиолетовую) и еще более легкую (красную), содержащую только ДНК с 14N, что подтверждало полу- консервативный характер репликации.
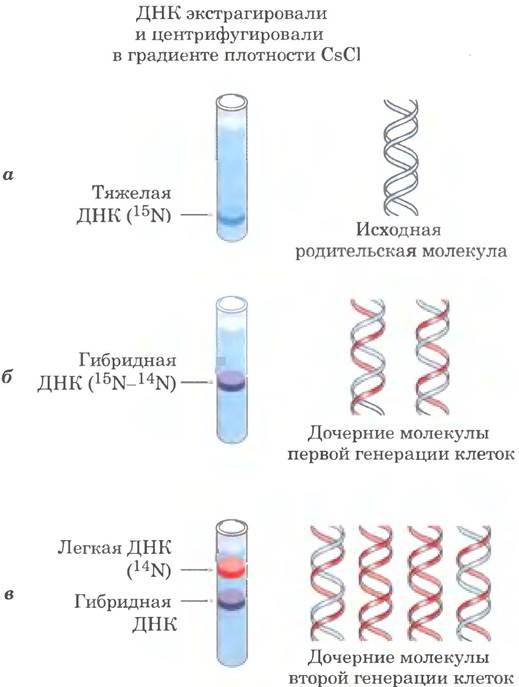
Клетки Е. coli, выращенные на среде с 15N, переносили на свежую питательную среду, содержащую только изотоп 14N, где клетки продолжали расти до тех пор, пока численность популяции не удваивалась. ДНК, выделенную из этой первой генерации клеток, центрифугировали в градиенте CsCl и наблюдали единственную полосу, положение которой свидетельствовало о том, что двухспиральная ДНК дочерних клеток представляет собой гибрид, содержащий одну новую цепь с 14N и одну родительскую цепь с 15N (рис. 25-2, б).
Представленные результаты опровергли альтернативную теорию консервативной репликации, согласно которой одна дочерняя молекула ДНК содержит две вновь синтезированные цепи, а другая дочерняя молекула — две родительские цепи; если бы эта теория была верна, в эксперименте Мезельсона -Сталя не было бы гибридной ДНК. Гипотеза полуконсервативной репликации дополнительно подтверждалась на следующем этапе эксперимента (рис. 25-2, в). После второго деления и удвоения клеток на среде с 14N из них была выделена ДНК, разделившаяся в градиенте хлорида цезия на две полосы: одна по плотности соответствовала легкой ДНК, а другая — гибридной ДНК. полученной после первого деления клеток.
Репликация начинается в точке начала репликации и обычно идет в двух направлениях.
После доказательства полуконсервативного механизма репликации возникло множество вопросов. Полностью ли раскручивается родительская ДНК до начала репликации каждой цепи? Начинается ли репликация в случайной или в какой-то особой точке? После инициации в какой-то точке репликация происходит в одном направлении или в двух?
В ранних исследованиях Джона Кэрнса, проведенным с помощью метода авторадиографии, удалось доказать, что репликация — высококоординированный процесс, в котором родительские цепи одновременно расплетаются и реплицируются. Кэрнс получил радиоактивную ДНК Е. coli, для чего культивировал эту бактерию на среде с меченным тритием (3Н) тимидином. ДНК была аккуратно выделена, покрыта слоем фотографической эмульсии и оставлена так на несколько недель; за это время радиоактивный тимидин оставил в фотоэмульсии «следы» — зерна серебра, создав фотоотпечаток молекулы ДНК. По этим отпечаткам видно, что интактная хромосома Е. coli представляет собой единую гигантскую кольцевую структуру длиной 1,7 мм. В радиоактивной ДНК, выделенной из клеток во время репликации, кроме того, была обнаружена дополнительная петля (рис. 25-3, а). Кэрнс сделал вывод, что, петля образуется в результате формирования двух радиоактивных дочерних цепей, каждая из которых комплементарна родительской пени. На одном или на обоих концах петли движется репликативная вилка, в которой родительская ДНК раскручивается, позволяя реплицироваться разделившимся цепям. Согласно полученным Кэрнсом данным, обе цепи ДНК реплицируются одновременно, а различные варианты его эксперимента (рис. 25-3, б) показали, что репликация бактериальных хромосом двунаправленная: на обоих концах петли расположены активные репликативные вилки.
Рис. 25-3. Выявление двунаправленного характера репликации ДНК. а — во время репликации кольцевой хромосомы образуется структура, напоминающая греческую букву тета (θ), так как обе нити реплицируются одновременно (новая нить показана красным цветом). б — репликация может происходить в одном или в обоих направлениях, что можно установить с помощью авторадиографии: при внесении 3Н на короткий период непосредственно перед прекращением реакции метка (отмечена красным) обнаруживается в одной или в двух репликативных вилках соответственно. Так была продемонстрирована двунаправленность репликации у Е. coli, Bacillus subtilis и других бактерий. На авторадиограмме показан репликационный «глазок» в ДНК В. subtilis. Самая большая плотность зерен серебра (стрелки) наблюдается именно в тех двух местах, где происходит репликация. Не подвергшаяся репликации часть хромосомы, находящаяся вне глазка, не содержит метки и поэтому невидима.
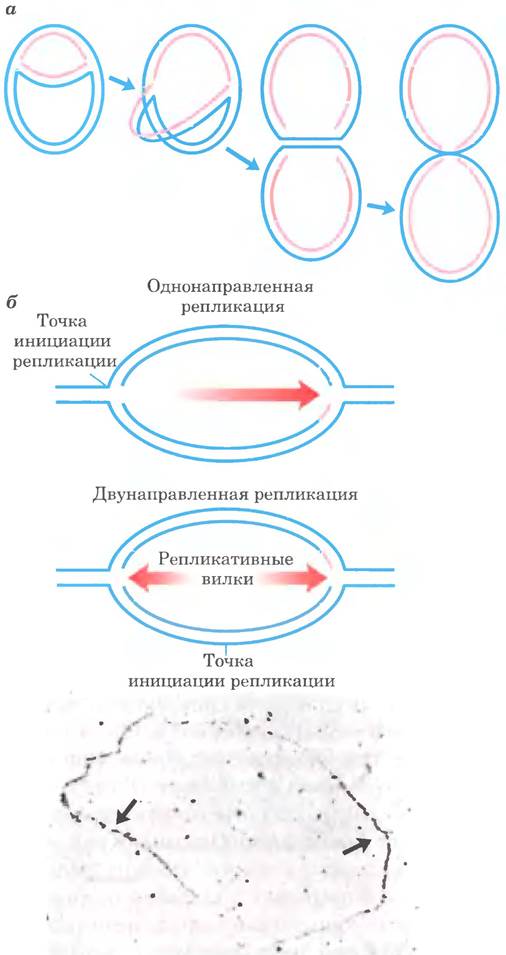
Чтобы выяснить, образуются ли репликативные вилки в каком-то определенном участке ДНК, необходимо было установить по всей длине молекулы некие реперные точки. Это было сделано с помощью метода денатурирующего картирования, разработанного Россом Инманом с коллегами. С помощью хромосомы бактериофага λ длиной 48 502 п. и. было показано, что можно вызвать избирательную денатурирацию ДНК в последовательностях с необычно высоким содержанием пар А = Т, получая воспроизводимую картину одноцепочечных глазков (см. рис. 8-28 в т. 1). Таким же образом можно частично денатурировать выделенную ДНК, содержащую репликативные петли. Это позволяет измерить и картировать положение и движение репликативных вилок, используя денатурированные области в качестве реперных точек. С помощью данного метода было показано, что в этой системе репликативные петли всегда возникают в особых точках, которые были названы точками начала репликации (ориджинами). Получил подтверждение и выявленный ранее двунаправленный характер репликации. У кольцевых молекул ДНК две репликативные вилки встречаются в точке, расположенной напротив точки начала репликации. Специфические точки начала репликации с тех пор были идентифицированы и охарактеризованы у бактерий и низших эукариот.
Синтез ДНК наполовину прерывистый и проходит в направлении 5'—> 3'.
Новая цепь ДНК всегда синтезируется в направлении 5' —> 3', т. е. приращение ДНК происходит со стороны свободной ОН-группы на 3'-конце (строение 5'- и 3'-концов цепи ДНК см. на рис. 8-7 в т. 1). Поскольку две цепи ДНК антипараллельны, цепь, служащая в качестве матрицы, считывается с 3’-конца в направлении к 5'-концу.
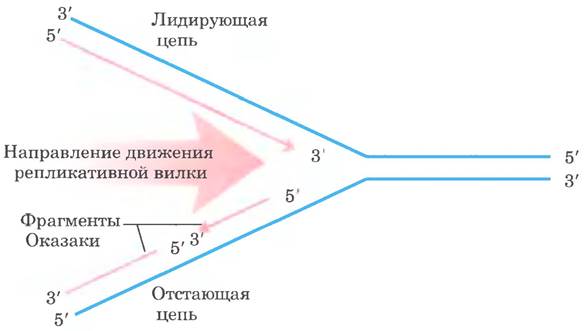
Но если синтез ДНК всегда происходит в направлении 5' —> 3', как могут обе цепи синтезироваться одновременно? Если обе цепи синтезируются непрерывно, пока движется репликативная вилка, одна цепь должна синтезироваться в направлении 3' —> 5'. Эту проблему в 1960-х гг. разрешил Рейджи Оказаки с коллегами. Они обнаружили, что одна из новых цепей ДНК синтезируется в виде коротких отрезков, которые теперь называются фрагментами Оказаки. В результате их исследований стало ясно, что одна цепь синтезируется непрерывно, а вторая — прерывисто (рис. 25-4). Направление синтеза непрерывной, или лидирующей, цепи совпадает с направлением движения репликативной вилки. Направление синтеза прерывистой, или отстающей, цепи противоположно направлению движения вилки. Фрагменты Оказаки различаются по длине от нескольких сотен до нескольких тысяч нуклеотидов в зависимости от типа клеток. Ниже мы увидим, что синтез лидирующей и отстающей цепей четко координируется.
Рис. 25-4. Различия цепей ДНК на репликативной вилке. Новая цепь ДНК (красная) всегда синтезируется в направлении 5' —> 3'. Матрица считывается в противоположном направлении. 3' —> 5'. Лидирующая цепь непрерывно синтезируется по направлению движения репликативной вилки. Другая цепь, отстающая, синтезируется прерывисто в виде коротких отрезков (фрагментов Оказаки) в направлении, противоположном тому, в котором движется репликативная вилка. Фрагменты Оказаки сшиваются ДНК-лигазой. У бактерий длина фрагментов Оказаки составляет примерно от 1000 до 2000 нуклеотидов. В клетках эукариот они короче: от 150 до 200 нуклеотидов.
ДНК разрушается нуклеазами
Чтобы объяснить энзимологию процесса репликации ДНК, сначала рассмотрим ферменты, которые разрушают, а не синтезируют ДНК. Эти ферменты называют нуклеазами или ДНКазами, если они обладают большей специфичностью к ДНК, чем к РНК. Каждая клетка содержит несколько разных нуклеаз, принадлежащих к двум большим классам: экзонуклеазы и эндонуклеазы. Экзонуклеазы расщепляют нуклеиновые кислоты с одного конца молекулы. Многие из них работают только в направлении 5' —> 3' или только в направлении 3' —> 5', удаляя нуклеотиды либо с 5'-конца, либо с 3'-конца одной цепи двухцепочечной молекулы нуклеиновой кислоты или одноцепочечной ДНК. Эндонуклеазы начинают расщепление нуклеиновой кислоты со специфических внутренних сайтов, расщепляя цепь на более мелкие фрагменты. Некоторые экзонуклеазы и эндонуклеазы расщепляют только одноцепочечные ДНК. Существует несколько важных классов эндонуклеаз, которые разрушают только определенные нуклеотидные последовательности (например, эндонуклеазы рестрикции, которые имеют очень большое значение в биотехнологии; см. гл. 9, рис. 9-2 вт. 1). В этой и в последующих главах мы встретим много типов нуклеаз.
ДНК синтезируется ДНК-полимеразами
Поиск фермента, который может синтезировать ДНК, начался в 1955 г. Артуру Корнбергу с коллегами удалось очистить и охарактеризовать ДНК-полимеразу из клеток Е. coli. Этот фермент, состоящий из одного полипептида, теперь называют ДНК-полимеразой I (Мr = 103 000; кодируется геном роlА). Гораздо позже исследователи обнаружили, что Е. coli содержит еще как минимум четыре другие ДНК-полимеразы, описанные ниже.

При детальном изучении ДНК-полимеразы 1 были установлены общие для всех ДНК-полимераз характеристики процесса синтеза ДНК. В основной реакции происходит перенос фосфорильных групп. При этом 3'-гидроксильная группа нуклеотида на 3'-конце растущей цепи выступает в роли нуклеофила, атакующего α-фосфор присоединяющегося дезоксинуклеозид-5'-трифосфата (рис. 25-5). В результате высвобождается неорганический пирофосфат. Основная реакция выглядит следующим образом:
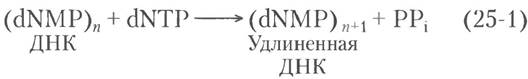
где dNMP и dNTP - дезоксинуклеозид-5v-монофосфат и дезоксинуклеозид-5'-трифосфат соответственно. Реакция происходит с минимальным изменением свободной энергии, поскольку одна фосфодиэфирная связь образуется из менее стабильного фосфоангидрида. Нековалентные стэкинговые взаимодействия оснований и их спаривание обеспечивают дополнительную стабилизацию удлиняющейся ДНК по сравнению со свободными нуклеотидами. Кроме того, синтез ДНК приводит к высвобождению в клетке 19 кДж/моль энергии в результате последующего гидролиза пирофосфата под действием фермента пирофосфатазы (с. 38 в т. 2).
Рис. 25-5. МЕХАНИЗМ РЕАКЦИИ. Элонгация цепи ДНК. а — ДНК-полимераза I нуждается в одной неспаренной цепи в качестве матрицы и в праймере, несущем на 3'-конце свободную гидроксильную группу, к которой присоединяется новое нуклеотидное звено. Каждый присоединяющийся нуклеотид выбирается по принципу комплементарности по отношению к соответствующему нуклеотиду в матричной цепи. Продукт реакции вновь содержит свободную 3'-гидроксильную группу, позволяющую присоединять следующий нуклеотид. б — для катализа, вероятно, требуется два иона Мg2+, координированных с двумя фосфатными группами присоединяющегося нуклеозидтрифосфата и с тремя остатками Asp, два из которых консервативны у всех ДНК-полимераз. Ион Мg2+, изображенный справа, облегчает атаку 3'-гидроксильной группы праймера на α-фосфат нуклеозидтрифосфата другой ион Мg2+ способствует удалению пирофосфата. Оба иона стабилизируют структуру пятикоординационного переходного состояния. По схожему механизму действует и РНК-полимераза (см. рис. 26-1, б).
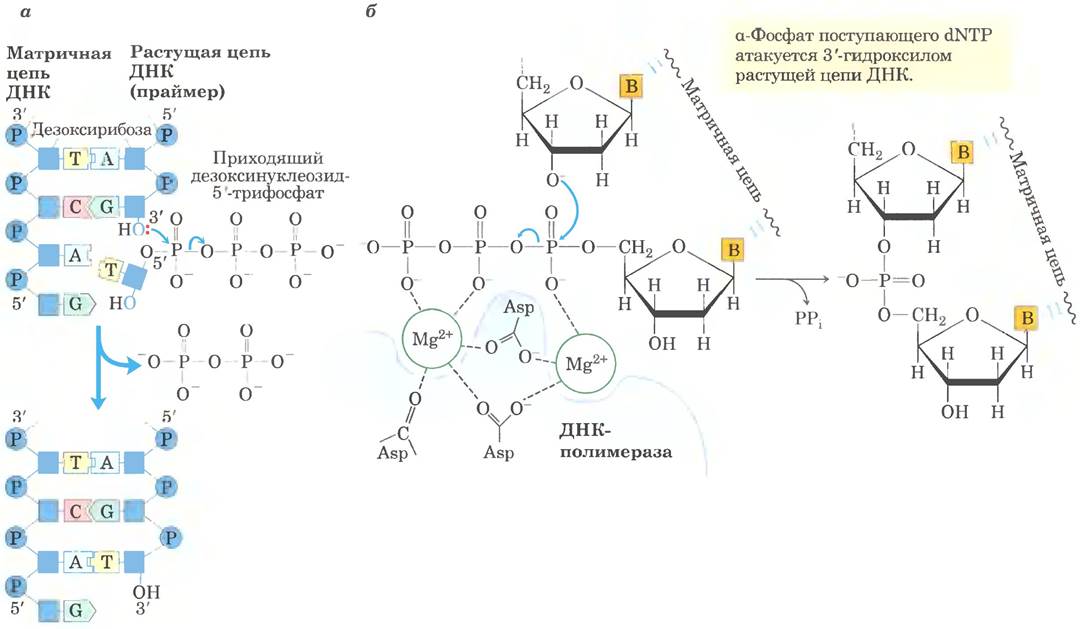
Еще в ранних исследованиях ДНК-полимеразы I удалось определить два основных условия, необходимых для полимеризации ДНК. Во-первых, все ДНК-полимеразы нуждаются в матрице. Реакция полимеризации происходит на матрице ДНК согласно правилам комплементарности оснований, установленным Уотсоном и Криком: если в матрице присутствует гуанин, к новой цепи присоединяется дезоксинуклеотид цитозина и т. д. Это открытие имеет очень важное значение не только потому, что оно объясняет химическую основу точной полуконсервативной репликации ДНК, но и потому, что это был первый пример использования матрицы для проведения реакции биосинтеза.
Во-вторых, полимеразы нуждаются в праймере. Праймер — это участок цепи (комплементарный матрице) со свободной З'-гидроксильной группой, к которой может присоединиться нуклеотид. Иначе говоря, перед началом синтеза часть новой цепи уже должна существовать: все ДНК-полимеразы могут присоединять нуклеотиды только к уже существующей цепи. Многие праймеры представляют собой олигонуклеотиды РНК, а не ДНК; специальные ферменты синтезируют праймеры там и тогда, где и когда они требуются.
После присоединения нуклеотида к растущей цепи ДНК ДНК-полимераза либо диссоциирует, либо продвигается вдоль матрицы и присоединяет следующий нуклеотид. Диссоциация и обратное присоединение полимеразы может ограничивать среднюю скорость полимеризации — процесс обычно протекает быстрее, если полимераза присоединяет нуклеотиды, не покидая матрицы. Среднее число нуклеотидов, присоединенных до диссоциации полимеразы, определяет ее процессивность. ДНК-полимеразы существенно различаются по процессивности; некоторые присоединяют всего несколько нуклеотидов до диссоциации, другие присоединяют многие тысячи.
Репликация — очень точный процесс
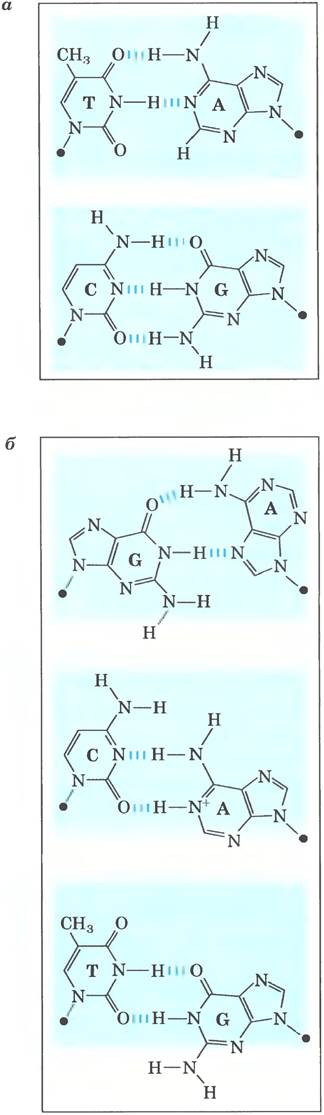
Репликация осуществляется с исключительной точностью. У Е. coli происходит всего одна ошибка на каждые 109-1010 присоединенных нуклеотидов. Для хромосомы Е. coli, размер которой составляет примерно 4,6 • 106 п. н., это означает, что за 1000-10 000 циклов репликации случается лишь одна ошибка. Во время полимеризации выбор между правильным и неправильным нуклеотидом осуществляется не только по специфичности водородных связей между комплементарными основаниями в парах, но также по геометрии стандартных А = Т и G = C пар оснований (рис. 25-6). В активном центре ДНК-полимеразы I могут разместиться только пары оснований с правильной геометрией. Неподходящий нуклеотид может образовать водородные связи с нуклеотидом в матрице, но такая пара обычно не попадает в активный центр фермента. Это позволяет отбраковать неправильное основание до того, как сформируется фосфодиэфирная связь.
Рис. 25-6. Значение геометрии пар оснований для точности репликации ДНК. а — стандартные пары оснований А = Т и G = C очень схожи по геометрии, и, если активный центр фермента может вместить одну пару (синяя рамка), то обычно он вмещает и другую, б — геометрия аномальных пар отличается, и они не помещаются в активном центре ДНК-полимеразы I.
Однако, чтобы обеспечить высокую степень точности репликации, одной только точности реакции полимеризации недостаточно. Измерения in vitro показали, что ДНК-полимераза вставляет один неправильный нуклеотид на 104 - 105 правильных нуклеотидов. Иногда эти ошибки возникают из-за того, что основание кратковременно находится в нехарактерной таутомерной форме (см. рис. 8-9 в т. 1), что позволяет ему образовать водородные связи не с тем партнером. Число ошибок in vivo уменьшается благодаря дополнительным ферментативным механизмам.
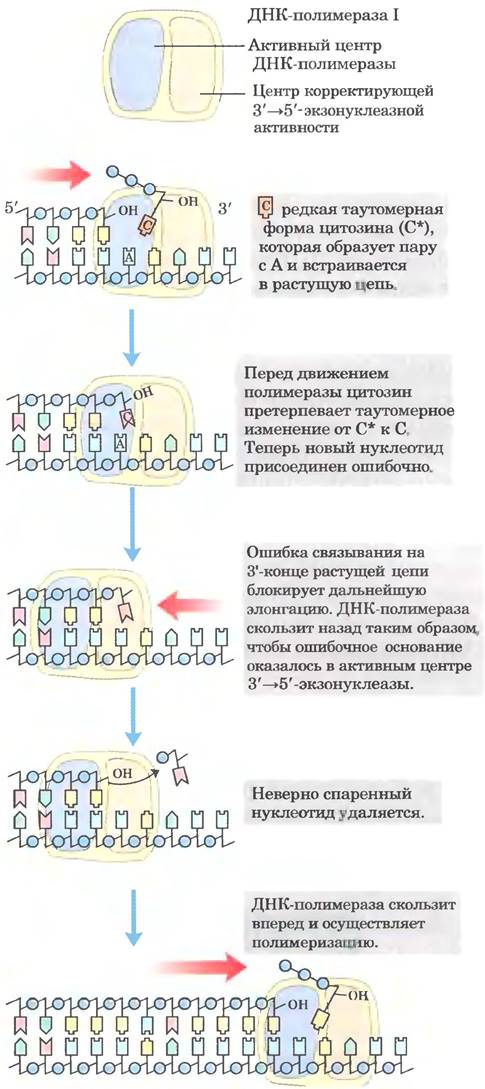
Один механизм сокращения числа ошибок свойствен практически всем ДНК- полимеразами и заключается в независимой 3' —> 5'-экзонуклеазной активности, благодаря которой каждый нуклеотид после присоединения проверяется дважды. Эта нуклеазная активность чрезвычайно чувствительна к аномальному спариванию оснований, что позволяет ферменту удалять ошибочно присоединенный нуклеотид (рис. 25-7). Если полимераза присоединила неправильный нуклеотид, происходит ингибирование ее перемещения к позиции, в которой должен присоединяться следующий нуклеотид. Эта пауза создает возможность для исправления ошибки. С помощью 3' —> 5'-экзонуклеазной активности полимераза удаляет неправильный нуклеотид и продолжает удлинение цепи. Данная активность, называемая корректирующей, не просто обращение полимеразной реакции (уравнение 25-1), ведь в ней не участвует пирофосфат. Полимеразную и корректирующую активности ДНК-полимеразы можно измерить независимо. Корректирующая активность дополнительно увеличивает точность реакции полимеризации в 102-103раз. У мономерной ДНК-полимеразы I полимеразная и корректирующая активности локализованы в разных активных центрах в одном и том же полипептиде.
Рис. 25-7. Пример исправления ошибки благодаря 3' —> 5'-экзонуклеазной активности ДНК-полимеразы I. При изучении структуры фермента было установлено, что при ориентации фермента по ходу движения ДНК- центр его экзонуклеазной активности локализован впереди центра полимеразной активности. Ошибочное спаривание (здесь ошибка С-А) препятствует транслокации ДНК-полимеразы I в следующее положение. Фермент скользит назад, исправляет ошибку благодаря своей 3' —> 5'-экзонуклеазной активности, а затем возобновляет полимеразную активность в направлении 5' —> 3'.
При тщательном подборе оснований в сочетании с исправлением ошибок ДНК-полимераза совершает примерно одну ошибку на 106-108 присоединенных оснований. В реальности измеряемая точность репликации Е. coli еще выше. Дополнительная точность обеспечивается независимой ферментативной системой, которая заменяет аномальные пары оснований, оставшиеся после репликации. Данный процесс исправления ошибок, наряду с другими процессами репарации ДНК, описан в разд. 25.2.
У Е. coli не менее пяти ДНК-полимераз
Более 90% ДНК-полимеразной активности в экстрактах Е. coli связано с функцией ДНК-полимеразы I. Однако вскоре после выделения этого фермента в 1955 г. стали накапливаться сведения о том, что он не может осуществлять репликацию крупной хромосомы Е. coli. Во-первых, скорость, с которой фермент присоединяет нуклеотиды (600 нуклеотидов/мин), не соответствует скорости движения репликативной вилки в бактериальной клетке (скорость движения репликативной вилки, как минимум, в 100 раз выше). Во- вторых, ДНК-полимераза I обладает относительно низкой процессивностью. В-третьих, генетические исследования показали, что в репликации участвуют многие гены и, следовательно, многие белки: очевидно, что ДНК-полимераза I действует не в одиночку. В-четвертых, и это особенно важно, в 1969 г. Джон Кэрнс выделил бактериальный штамм с поврежденным геном ДНК-полимеразы I, с которого синтезировался неактивный фермент. Хотя этот штамм был чрезвычайно чувствителен к повреждениям ДНК, тем не менее он был жизнеспособен!
Поиск других ДНК-полимераз привел к открытию в начале 1970-х гг. ДНК-полимеразы II и ДНК-полимеразы III у Е. coli. ДНК-полимераза II участвует в одном из механизмов репарации ДНК (разд. 25.3). ДНК-полимераза III — главный фермент репликации у Е. coli. Свойства трех ДНК-полимераз Е. coli сравниваются в табл. 25-1. ДНК-полимеразы IV и V, идентифицированные в 1999 г., принимают участие в необычном варианте репарации ДНК (разд. 25.2).
Таблица 25-1. Сопоставление ДНК-полимераз Е. coli
ДНК-полимер |
аза |
||
I |
II |
III |
|
Структурный гена |
polA |
роlB |
polC (dnaE) |
Субъединицы (количество разных типов) |
1 |
7 |
≥10 |
Относительная молекулярная масса (Mr) |
103 000 |
88 000б |
791 500 |
3' —> 5'-экзонуклеаза (коррекция ошибок) |
Да |
Да |
Да |
5' —> 3'-экзонуклеаза |
Да |
Нет |
Нет |
Скорость полимеризации (нуклеотиды/с) |
16-20 |
10 |
250- 1000 |
Процессивность (число нуклеотидов, присоединенных до диссоциации полимеразы) |
3-200 |
1500 |
≥500 000 |
а Для субъединичных ферментов назван ген, кодирующий субъединицу с полимеразной активностью. Ген dnаE в настоящее время называется polC.
б Только субъединица, отвечающая за полимеризацию. В составе ДНК-полимеразы II есть те же субъединицы, что и у ДНК-полимеразы III: (β-, y-, δ-, δ'-, χ- и Ψ-субъединицы (см. табл. 25-2).
ДНК-полимераза I, таким образом, не главный фермент в репликации; она выполняет функцию проверки и поддержания порядка во время репликации, рекомбинации и репарации. Особые функции полимеразы дополняются ее 5' —> 3'-экзо- нуклеазной активностью. Эта активность, отличная от корректирующей 3' —> 5'-экзонуклеазной активности (рис. 25-7), локализована в структурном домене, который может быть отщеплен от фермента в условиях мягкой обработкой протеазами. При удалении 5' —> 3'-экзонуклеазного домена остающийся фрагмент (Мr = 68 000), называемый большим фрагментом, или фрагментом Клёнова (рис. 25-8), сохраняет полимеразную и корректирующую активность. Интактная ДНК-полимераза I благодаря своей 5' —> 3'-экзонуклеазной активности в ходе так называемой ник-трансляции может заменить участок ДНК (или РНК), связанный с матричной цепью (рис. 25-9). Большинство других ДНК-полимераз не имеют 5' —> 3'-экзонуклеазной активности.
Рис. 25-8. Большой фрагмент ДНК-полимеразы I (фрагмент Клёнова). Эта полимераза широко распространена у бактерий. Фрагмент Клёнова, образующийся в результате протеолитической обработки полимеразы, сохраняет полимеразную и корректирующую активности. Представленный на рисунке фрагмент Клёнова получен из термофильной бактерии Bacillus stearothermophilus (PDB ID 3BDP). Активный центр полимеразы находится глубоко в щели около дальнего конца связанной ДНК (синяя; темно-синим цветом изображена матричная цепь).
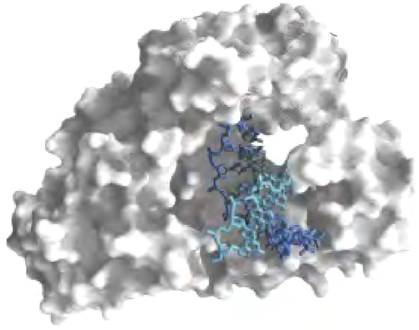
Рис. 25-9. Ник-трансляция. В ходе этого процесса участок РНК или ДНК, спаренный с матричной ДНК, одновременно разрушается под действием 5' —> 3'-экзонуклеазной активности ДНК-полимеразы I и заменяется на новый под действием полимеразной активности того же фермента. Эта функция реализуется в процессе репарации ДНК и при удалении РНК-праймеров в ходе репликации (оба процесса описаны ниже). Цепь нуклеиновой кислоты (ДНК или РНК), подлежащая удалению, показана зеленым, новая цепь — красным. Синтез ДНК начинается с ника (разрыва фосфодиэфирной связи, освобождающего 3'-гидроксильную и 5'-фосфатную группы). Полимераза I удлиняет нематричную цепь ДНК и перемещает ник вдоль ДНК. Этот процесс называется ник-трансляцией. В месте диссоциации ДНК-полимеразы ник сохраняется и позднее зашивается другим ферментом.
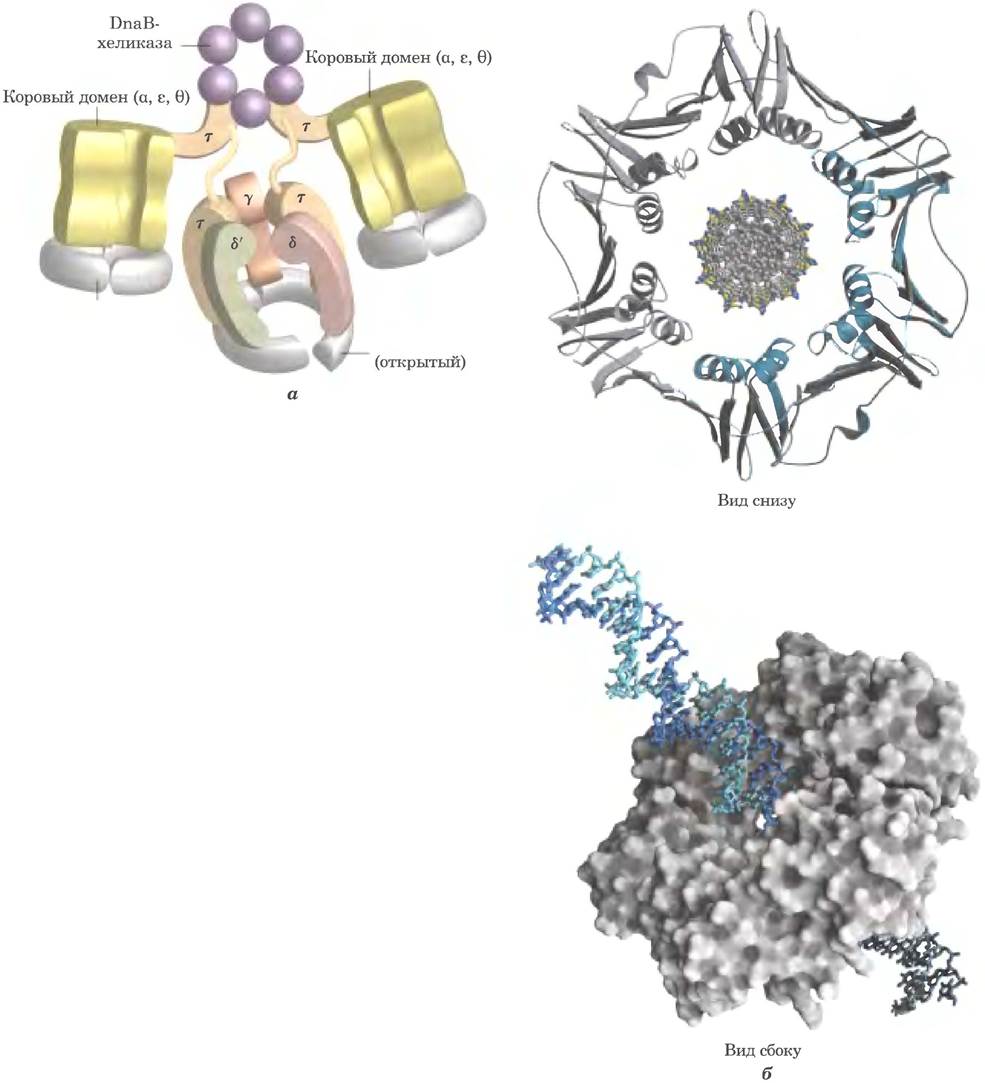
ДНК-полимераза III устроена намного сложнее, чем ДНК-полимераза I; она состоит из субъединиц десяти разных типов (табл. 25-2). Ее полимеразная и корректирующая активности связаны с α- и ε-субъединицами соответственно. При объединении θ-субъединицы с α- и ε-субъединицами образуется корфермент, который уже может осуществлять синтез ДНК, но с ограниченной процессивностью. Два кор-фермента полимеразы могут связываться друг с другом посредством еще одного субъединичного комплекса, называемого погрузчиком зажима, или y-комплексом, состоящим из пяти субъединиц четырех разных типов Ƭ2yδδ'. Кор-ферменты связаны через Ƭ-субъединицы. Две дополнительные единицы χ и Ψ, присоединены к погрузчику зажима. Полный набор из 13 белковых субъединиц (девяти разных типов) называется ДНК-полимеразой III* (рис. 25-10, а).
Таблица 25-2. Субъединицы ДНК-полимеразы III E. coli

а y-Субъединицу кодирует часть гена Ƭ-субъединицы, так что 66% Ƭ-субъединицы с N-конца совпадают по аминокислотной последовательности с y-субъединицей. y-Субъединица образуется в результате смещения рамки считывания при трансляции (см. рис. 27-9), что приводит к преждевременной остановке трансляции.
ДНК-полимераза III* способна полимеризовать ДНК, но с намного меньшей процессивностью, чем нужно для организованной репликации целой хромосомы. Необходимое увеличение процессивности обеспечивается присоединением четырех β-субъединиц с образованием полного фермента ДНК-полимеразы III. β-Субъединицы объединяются в пары и формируют структуры в форме бубликов, которые охватывают молекулы ДНК, как зажимы (рис. 25-10, б). Каждый димер связывается с кор-ферментом полимеразы III* (один димерный зажим на кор-фермент) и во время репликации скользит вдоль ДНК. Скользящий β-зажим предупреждает диссоциацию ДНК-полимеразы III из комплекса с ДНК, резко увеличивая процессивность до 500 000 нуклеотидов и более (табл. 25-1).
Рис. 25-10. ДНК-полимераза III. а — строение бактериальной ДНК-полимеразы III. Два центральных домена (кор-ферменты), состоящие из субъединиц α, ε и θ, связаны с y-комплексом (погрузчиком зажима), состоящим из пяти субъединиц Ƭ2yδδ'. Субъединицы y и Ƭ кодируются одним и тем же геном. Субъединица у представляет собой укороченную субъединицу Ƭ: субъединица Ƭ содержит домен, идентичный у, и дополнительный сегмент, взаимодействующий с кор-ферментом. Две другие субъединицы χ и Ψ (не показаны) ДНК-полимеразы III* также связаны с y-комплексом. Два β-зажима присоединяются к двум кор-ферментам; каждый зажим — это димер β-субъединицы. Комплекс взаимодействует с DnaB- хеликазой через Ƭ-субъединицу. б — две β-субъединицы полимеразы III Е. coli образуют зажим в виде кольца, которое окружает ДНК. Зажим скользит по молекуле ДНК, увеличивая процессивность холофермента ДНК-полимеразы III до 500 000 нуклеотидов и более, не позволяя ДНК диссоциировать. Вид снизу демонстрирует две β-субъединицы, изображенные в виде лент голубого и серого цвета, окружающих СРК-модель молекулы ДНК. Вид сбоку демонстрирует контур поверхности β-субъединиц (серого цвета), которые окружают двойную спираль ДНК (голубой и синий цвет), изображенную в виде стрежневой модели (по PDB ID 2PОL).
В репликации ДНК участвует множество ферментов и белковых факторов
Для репликации Е. coli нужна не только ДНК-полимераза, но также не менее 20 различных ферментов и белков, каждый из которых выполняет определенную задачу. Весь этот комплекс был назван ДНК-репликативной системой, или реплисомой. Сложность ферментативного аппарата репликации связана со структурой ДНК и с требованиями высокой точности синтеза. Основные классы ферментов репликации перечислены ниже в соответствии с теми функциями, которые они выполняют.
Чтобы обеспечить доступ к цепям ДНК, которые служат в качестве матрицы, две родительские цепи нужно разделить. Обычно этот процесс осуществляют хеликазы — ферменты, которые движутся вдоль ДНК и разделяют ее цепи, затрачивая при этом энергию АТР. Разделение цепей создает топологическое напряжение в спиральной структуре ДНК (см. рис. 24-12), которое снимается топоизомеразами. Разделенные цепи стабилизируются ДНК-связывающими белками. Как уже было сказано, прежде чем ДНК-полимеразы смогут начать синтез ДНК, на матрице должны находиться праймеры — обычно это короткие сегменты РНК, синтезированные ферментами праймазами. Позднее РНК-праймеры удаляются и заменяются на ДНК; у Е. coli это действие — одна из основных функций ДНК-полимеразы I. После вырезания РНК-праймера и встраивания ДНК в последовательности ДНК сохраняется ник в форме разорванной фосфодиэфирной связи. Эти ники зашивают ДНК- лигазы. Все перечисленные процессы нуждаются в координации и регуляции, которые хорошо исследованы в системе Е. coli.
Репликация хромосомы E. coli происходит постадийно
В синтезе молекулы ДНК можно выделить три стадии: инициация, элонгация и терминация, которые различаются как по химической сути, так и по составу ферментов. В этой и двух последующих главах рассматриваются именно эти три общие стадии синтеза крупных информационных полимеров ДНК, РНК и белков, а также специфические особенности каждого биосинтетического пути. Описываемые далее события базируются на информации, первоначально полученной в экспериментах in vitro на очищенных белках Е. coli, хотя принципы репликации весьма консервативны во всех системах.
Инициация.
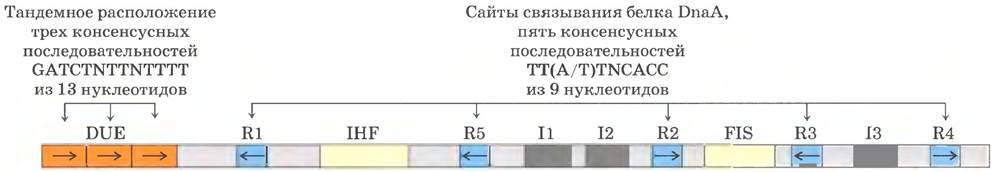
Точка начала репликации Е. coli (oriC) состоит из 245 п. н.; она содержит элементы последовательности, консервативные во многих бактериальных точках начала репликации. Стандартный набор консервативных последовательностей показан на рис. 25-11. Наибольший интерес представляют два типа последовательностей: пять повторов размером по 9 п. н. (R-сайты) - участки связывания ключевого инициаторного белка DnaA, а также А = Т-богатый участок, называемый ДНК-расплетающим элементом DUE (от англ. DNA unwinding element). Кроме того, существуют три дополнительных участка связывания DnaA (I -сайты) и участки связывания белков IHF (от англ. integration hose factor) и FIS (от англ. factor for inversion stimulation). Эти два белка необходимы для некоторых реакций рекомбинации, описанных ниже в данной главе, а их названия отражают их функции. Еще один ДНК-связывающий белок HU (гистоноподобный бактериальный белок, ранее называвшийся фактором U) также участвует в процессе, но не имеет специфического участка связывания.
Рис. 25-11. Расположение последовательностей в точке начала репликации Е. coli (oriC). Показаны консенсусные последовательности (см. с. 156 в т. 1) ключевых повторов. N обозначает любой из четырех нуклеотидов. Горизонтальные стрелки показывают ориентацию нуклеотидных последовательностей (стрелки слева направо указывают на последовательность в верхней цепи, а справа налево — в нижней). Участки FIS и IHF — сайты связывания белков, описанных в тексте. С R-сайтами связан белок DnaA. Сайты I — дополнительные участки связывания DnaA (у них другие последовательности). DnaA взаимодействует с ними только в комплексе с АТР.
В инициации репликации участвуют по меньшей мере 10 разных ферментов и белков (см. табл. 25-3). Они расплетают спираль ДHК в точке начала репликации и собирают препраймирующий комплекс для последующих реакций. Ключевым элементом в процессе инициации выступает белок DnaA — входит в белковое семейство ААА+ АТРаз (от англ. ATPases associated with diverse cellular activities). Многие AAA+ АТРазы, включая DnaA, образуют олигомерные структуры и сравнительно медленно гидролизуют АТР. Гидролиз АТР служит переключателем, опосредующим переходы одной формы белка в другую. В случае DnaA ATP-связанная форма активна, а ADP-связанная форма неактивна.
Восемь молекул белка DnaA, каждая из которых связана с АТР, образуют спиральный комплекс, охватывающий R- и I-сайты в oriC (рис. 25-12). DnaA имеет более высокое сродство к R-сайтам, чем к 1-сайтам, и этот белок связывается с R-сайтами одинаково хорошо как в АТР-, так и в ADP-форме. Напротив, 1-сайты, в которых связывается только ATP-связанный DnaA, позволяют различать активную и неактивную форму DnaA. Молекула ДНК плотно закручивается в правую сторону вокруг этого комплекса, образуя положительную суперспираль (см. гл. 24). Возникшее в соседних участках ДНК напряжение вызывает денатурацию А = Т-богатого участка DUE. Кроме того, образующийся в точке начала репликации комплекс также содержит несколько ДНК-связываюших белков - HU, IHF и FIS, которые облегчают изгиб ДНК.
Рис. 25-12. Модель инициации репликации в точке начала репликации Е. coli (оri'С). Восемь связанных с АТР молекул белка DnaA присоединяются к точке начала репликации в R- и I-сайтах (см. рис. 25-11). ДНК закручивается вокруг этого комплекса с образованием правозакрученной спирали. Богатый остатками А = Т участок DUE денатурирует из-за напряжения, возникающего в цепи в результате связывания DnaA. Образованию спирального комплекса с DnaA способствуют белки HU, IHF и FIS (здесь не показаны, так как подробно их структура и роль еще не определены). Гексамеры белка DnaB связываются с каждой цепью ДНК с помощью белка DnaC. Благодаря своей хеликазной активности белок DnaB раскручивает ДНК, подготавливая ее для посадки праймера и синтеза ДНК.
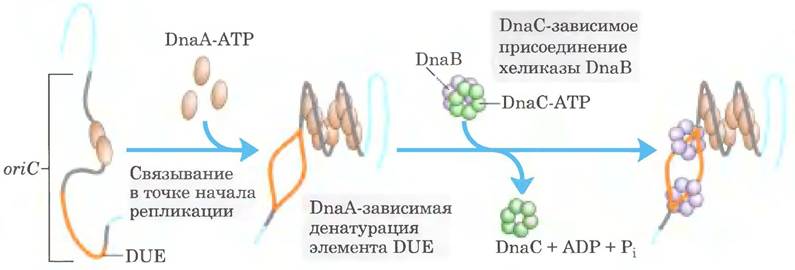
Затем другая ААА+ АТРаза, DnaC, загружает белок DnaB на разделенные цепи ДНК в денатурированном участке. Гексамер, состоящий из молекул DnaC, каждая из которых связана с АТР, образует прочный комплекс с гексамерной кольцевой DnaB-хеликазой. При взаимодействии DnaC с DnaB кольцо DnaB раскрывается, чему также способствует дальнейшее взаимодействие между DnaB и DnaA. Два кольцевых гексамера DnaB связываются с DUE — по одному на каждую нить ДНК. Связанная с DnaC молекула АТР гидролизуется, что приводит к высвобождению DnaC, a DnaB остается связанным с ДНК.
Присоединение хеликазы DnaB — ключевой момент в инициации репликации. Далее репликативная хеликаза DnaB перемещается вдоль одноцепочечной ДНК в направлении 5' —> 3', раскручивая ДНК по мере продвижения. Таким образом. связанные с двумя пенями ДНК молекулы хеликазы DnaB перемещаются в противоположных направлениях, создавая две репликативные вилки. Все другие белки репликативной вилки также напрямую или опосредованно связаны с DnaB. Холофермент ДНК-полимеразы III связан через Ʈ-субъединицы. Другие взаимодействия DnaB описаны ниже. В начале репликации, когда нити ДНК разделяются в области репликативной вилки, с ними связываются, стабилизируя их, многочисленные молекулы белка, связывающегося с одноцепочечной ДНК (single-stranded DNA-binding protein, SSB), а ДНК-гираза (ДНК- топоизомераза II) снимает топологическое напряжение, возникающее впереди вилки из-за раскручивания ДНК.
Инициация — единственная фаза репликации ДНК, про которую известно, что она регулируется, притом таким образом, что репликация происходит только один раз за клеточный цикл. Механизм регуляции пока еще точно не установлен. но в ходе генетических и биохимических исследований было выявлено несколько независимых элементов регуляции.
Как только с ДНК связывается ДНК-полимераза III, а также β-субъединицы (что свидетельствует о завершении фазы инициации), с β-субъединицами связывается белок Hda, который взаимодействует с DnaA и стимулирует гидролиз связанного с ним АТР. Белок Hda (название этого фермента означает «гомологичный DnaA») — еще одна ААА+ АТРаза, родственная DnaA. Гидролиз АТР приводит к разборке комплекса DnaA в точке начала репликации. Медленное высвобождение ADP и связывание новых молекул АТР замыкает цикл превращений между неактивной (со связанным ADP) и активной (со связанным АТР) формами белка, который длится от 20 до 40 мин.
На ход инициации репликации влияет метилирование ДНК и взаимодействия с бактериальной плазматической мембраной. ДНК в области оriС метилируется Dam-метилазой (от англ. DNA adenine methylation.; табл. 25-3), которая вводит метальную группу по положению N6 в остатке аденина в палиндромной последовательности (5’) GATC. Точка начала репликации оriС у Е. coli содержит особенно много повторов GATC — их 11 в последовательности из 245 п. н. тогда как в среднем во всей хромосоме Е. coli этот повтор встречается одни раз на 256 п. н.
Таблица 25-3. Белки, необходимые для инициации репликации в точке начала репликации E. coli
Белок |
Мr |
Число субъединиц |
Функция |
Белок DnaA |
52 000 |
1 |
Распознает последовательности в точке начала репликации; раскрывает цепи ДНК в специальных сайтах точки начала репликации |
Белок DnaB (хеликаза) |
300 000 |
6* |
Раскручивает ДНК |
Белок DnaC |
29 000 |
6* |
Связывает DnaB в точке начала репликации |
HU |
19 000 |
2 |
Гистоноподобный ДНК-связывающий белок; стимулирует инициацию |
FIS |
22 500 |
2* |
ДНК-связывающий белок; стимулирует инициацию |
IHF |
22 000 |
2 |
ДНК-связывающий белок; стимулирует инициацию |
Праймаза (белок DnaG) |
60 000 |
1 |
Синтезирует РНК-праймеры |
Белок, связывающий одноцепочечную ДНК (SSB) |
75 600 |
4* |
Связывает одноцепочечную ДНК |
ДНК-гираза (ДНК-топоизомераза II) |
400 000 |
4 |
Ослабляет торсионное напряжение, вызываемое раскручиванием ДНК |
Dam-метилаза |
32 000 |
1 |
Метилирует последовательность (5')GATC в oriС |
* В данном случае субъединицы одинаковые.
Непосредственно после репликации ДНК оказывается наполовину метилированной; в родительских цепях последовательности оriС метилированы, а во вновь синтезированных — нет. Эти наполовину метилированные последовательности оriС изолированы за счет взаимодействия с плазматической мембраной (механизм неизвестен) и связывания с белком SeqA. Через некоторое время последовательность оriС отделяется от плазматической мембраны, SeqA диссоциирует, и Dam-метилаза полностью метилирует ДНК до того, как она вновь связывает DnaA в новом раунде репликации.
Элонгация.
Фаза элонгации репликации включает две связанные между собой операции: синтез лидирующей цепи и синтез отстающей цепи. Для синтеза обеих цепей важно присутствие в репликатвной вилке нескольких ферментов. Сначала исходная ДНК раскручивается ДНК-хеликазой, а возникающее топологическое напряжение снимается топоизомеразами. Затем белок SSB стабилизирует каждую отдельную цепь. С этого момента синтез лидирующей цени и синтез отстающей цепи кардинально различаются.
Синтез лидирующей цепи происходит проще: он начинается с синтеза праймазой (белок DnaG) короткого (от 10 до 60 нуклеотидов) РНК- праймера в точке начала репликации. Для осуществления данной реакции DnaG взаимодействует с хеликазой DnaB. причем праймер синтезируется в направлении, которое противоположно направлению движения хеликазы DnaB. На самом деле хеликаза DnaB перемещается вдоль цепи, которая становится отстающей при синтезе ДНК. Однако первый праймер, синтезированный при первом взаимодействии DnaG-DnaB, служит для запуска синтеза лидирующей цепи ДНК в противоположном направлении. Дезоксирибонуклеотиды добавляются к этому праймеру ДНК-полимеразой III, связанной с DnaB-хеликазой, расположенной на противоположной цепи ДНК. Синтез лидирующей цепи происходит непрерывно со скоростью, соответствующей скорости раскручивания ДНК в репликативной вилке.
Как уже было отмечено, отстающая цепь синтезируется в виде коротких фрагментов Оказаки (рис. 25-13, а). Сначала праймаза синтезирует РНК-праймер. Затем, как и в процессе синтеза лидирующей цепи, ДНК-полимераза III связывается с праймером и присоединяет дезоксирибонуклеотиды (рис. 25-13, б). На этом этапе синтез каждого фрагмента Оказаки кажется простым, но на самом деле это весьма сложный процесс. Сложность заключается в координации синтезов лидирующей и отстающей цепей: обе цепи синтезируются одним асимметричным димером ДНК-полимеразы III, что достигается при образовании петли ДНК из отстающей цепи (рис. 25-14), сближающей две точки полимеризации.
Рис. 25-13. Синтез фрагментов Оказаки, а — на определенном расстоянии праймаза синтезирует РНК-праймер для нового фрагмента Оказаки. Если рассматривать две параллельные матричные цепи, синтез отстающей цепи формально происходит в направлении, противоположном направлению движения вилки, б — каждый праймер удлиняется ДНК-полимеразой III. в — синтез ДНК продолжается до тех пор, пока полимераза не достигнет праймера предыдущего фрагмента Оказаки. Для возобновления процесса вблизи репликативной вилки синтезируется новый праймер.
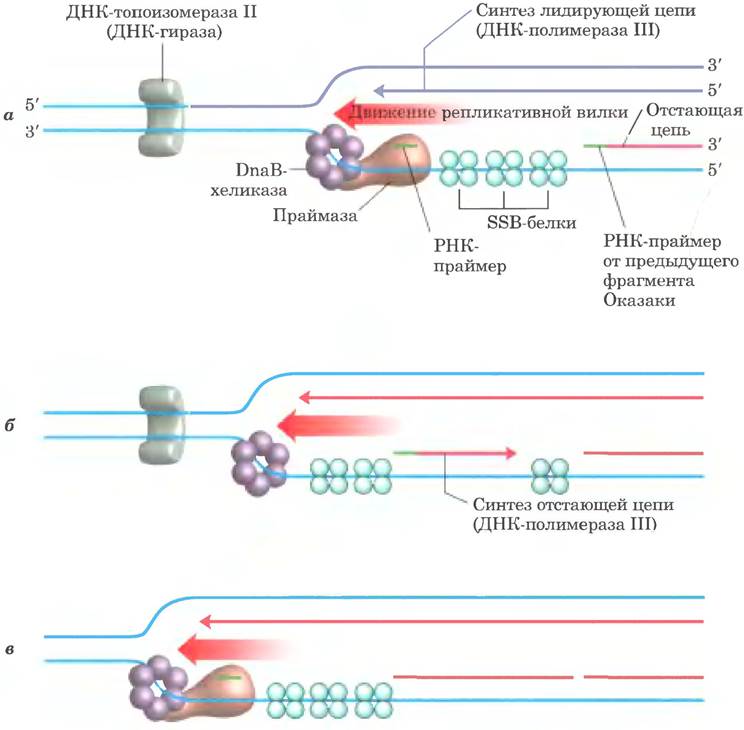
Синтез фрагментов Оказаки на отстающей цепи происходит с участием удивительного ферментативного аппарата. DnaB-хеликаза и DnaG- праймаза образуют отдельный функциональный блок, праймосому, в составе репликативного комплекса. ДНК-полимераза III использует один набор кор-ферментов (коровая полимераза) для непрерывного синтеза лидирующей цепи, в то время как другой набор кор-ферментов осуществляет циклы синтеза фрагментов Оказаки на петле отстающей цепи. DnaB-хеликаза, находящаяся перед ДНК-полимеразой III, раскручивает ДНК в репликативной вилке (рис. 25-14, а), продвигаясь вдоль матрицы отстающей цепи в направлении 5' —> 3'. ДНК-праймаза время от времени связывается с DnaB-хеликазой и синтезирует короткий РНК-праймер (рис. 25-14, б). Затем погрузчик зажима ДНК-полимеразы III закрепляет на праймере новый скользящий β-зажим (рис. 25-14, в). Когда синтез фрагмента Оказаки заканчивается, репликация приостанавливается, кор-фермент ДНК-полимеразы III отсоединяется от одного зажима (а также от законченного фрагмента Оказаки) и связывается с новым зажимом (рис. 25-14, г, б). Это инициирует синтез нового фрагмента Оказаки. Как отмечалось ранее, весь комплекс, ответственный за координированный синтез ДНК в репликатиной вилке, называют реплисомой. Белки, задействованные в репликативной вилке, перечислены в табл. 25-4.
Таблица 25-4. Белки реплисомы E. coli
Белок |
Мr |
Число субъединиц |
Функция |
SSB |
75 600 |
4 |
Связывание с одноцепочечной ДНК |
Белок DnaB (хеликаза) |
300 000 |
6 |
Раскручивание ДНК; компонент праймосомы |
Праймаза (белок DnaG) |
60 000 |
1 |
Синтез РНК-праймера; компонент праймосомы |
ДНК-полимераза III |
791 500 |
17 |
Элонгация новой цепи |
ДНК-полимераза I |
103 000 |
1 |
Заполнение брешей; вырезание праймеров |
ДНК-лигаза |
74 000 |
1 |
Лигирование |
ДНК-гираза (ДНК-топоизомераза II) |
400 000 |
4 |
Суперскручивание |
Рис. 25-14. Синтез ДНК на лидирующей и отстающей цепях. События, происходящие в репликативной вилке, координируются одним димером ДНК-полимеразы III в комплексе с DnaB-хеликазой. Здесь показан уже идущий процесс репликации (стадии от а до д обсуждаются в тексте). Отстающая цепь изогнута петлей, так что синтез ДНК происходит одновременно на лидирующей и на отстающей матрице. Красные стрелки указывают 3'-концы двух новых цепей и направление синтеза ДНК, толстые черные стрелки — направление движения родительской ДНК через комплекс. На отстающей цепи начинает синтезироваться фрагмент Оказаки. Цветовое обозначение субъединиц и функции погрузчика зажима объясняются на рис. 25-15.
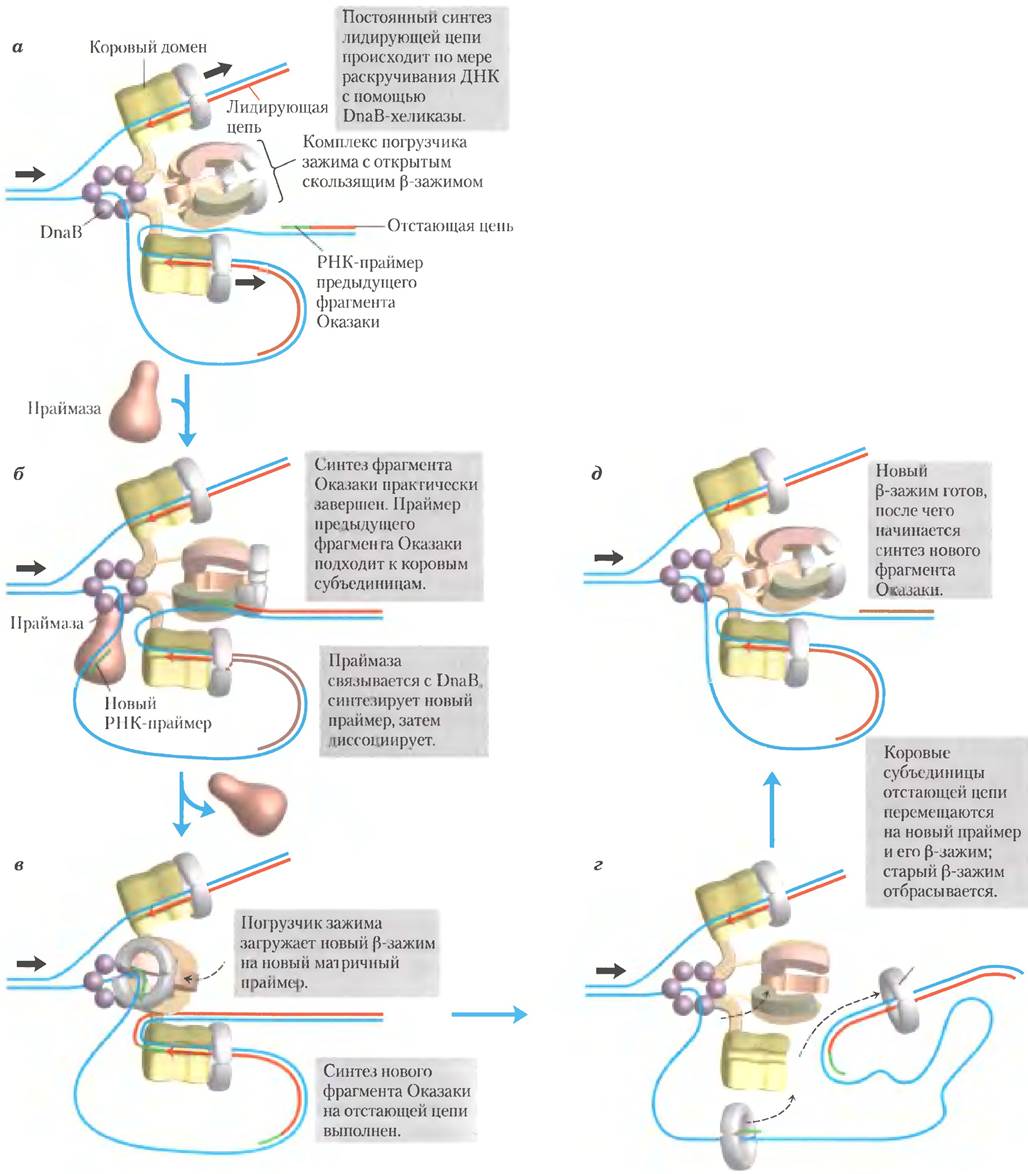
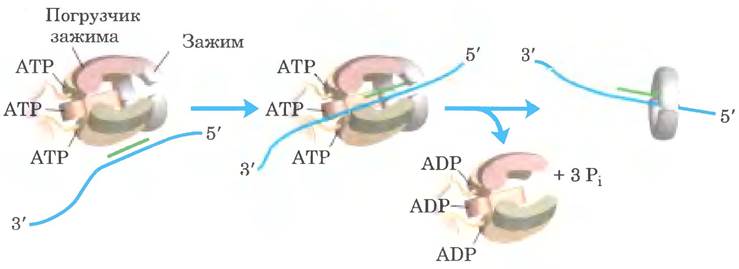
Погрузчик зажима ДНК-полимеразы III. состоящий из частей двух Ʈ-субъединиц, а также из y, δ и δ'-субъединиц, функционирует, кроме того, как ААА+ АТРаза. Этот комплекс связывается с АТР и с новым скользящим β-зажимом. Связывание приводит к возникновению напряжения в димерном зажиме, в результате чего на границе одной из субъединиц открывается кольцо (рис. 25-15). Отстающая цепь с только что присоединенным праймером проскальзывает в кольцо через образующийся разрыв. Затем погрузчик зажима гидролизует АТР, высвобождая скользящий (3-зажим и позволяя ему сомкнуться вокруг ДНК.
Рис. 25-15. Комплекс погрузчика зажима ДНК-полимеразы III. Комплекс образован пятью субъединицами: субъединица y, δ и δ', а также N-концевыми доменами обеих Ʈ-субъединиц (см. рис. 25-10). Комплекс связывает три молекулы АТР и димерный β-зажим. Это связывание приводит к открыванию β-зажима на границе одной из двух субъединиц. Гидролиз связанного АТР позволяет β-зажиму вновь замкнуться вокруг ДНК.
Реплисома осуществляет синтез ДНК быстро, присоединяя примерно по 1000 нуклеотидов в секунду к каждой цепи (лидирующей и отстающей). После завершения сборки одного фрагмента Оказаки его РНК-праймер удаляется и замещается последовательностью ДНК с помощью ДНК-полимеразы I, а оставшийся ник ликвидируется ДНК-лигазой (рис. 25-16).
Рис. 25-16. Заключительные стадии синтеза фрагментов отстающей цепи. ДНК-полимераза I благодаря своей 5' —> 3'-экзонуклеазной активности удаляет РНК- праймеры из отстающей цепи и замещает их на ДНК. Оставшийся ник (разрыв) ликвидирует ДНК-лигаза. Роль АТР и NAD+ показана на рис. 25-17.
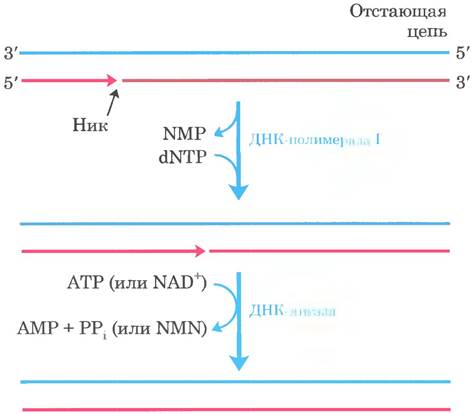
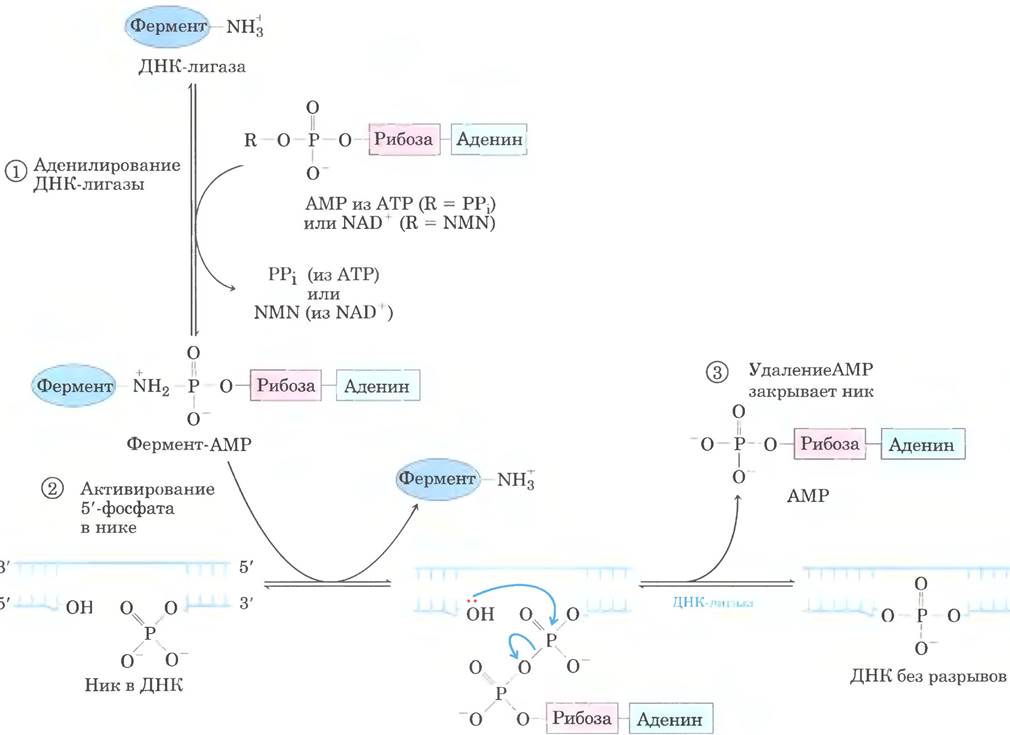
ДНК-лигаза катализирует образование фосфодиэфирной связи между 3’-гидроксильной группой на конце одной цепи ДНК и 5’-фосфатом на конце другой цепи. Фосфат должен быть предварительно активирован путем аденилирования. ДНК-лигазы, выделенные из вирусов и эукариот, используют для этой цели АТР. ДНК-лигазы бактерий (рис. 25-17) отличаются тем, что в качестве источника АМР они используют NAD+- кофактор, который обычно участвует в реакциях с переносом гидрид-иона (см. рис. 13-24). ДНК- лигаза еще один фермент метаболизма ДНК, который стал важным инструментом в экспериментах с рекомбинантными ДНК (см. рис. 9-1).
Рис. 25-17. Механизм реакции. Действие ДНК-лигазы. На каждой из трех стадий одна фосфодиэфирная связь образуется за счет другой. Стадии ① и ② приводят к активации 5'-фосфатной группы в нике. АМР сначала переносится на остаток лизина в молекуле фермента, а затем на 5'-фосфат в нике. На стадии ③ 3'-гидроксильная группа атакует этот фосфат и вытесняет АМР, образуя фосфодиэфирную связь и закрывая тем самым разрыв. У Е. coli АМР в реакции ДНК-лигазы происходит из NAD+. ДНК- лигазы, выделенные из некоторых вирусных и эукариотических источников, используют АТР, а не NAD+, и на стадии ① они высвобождают пирофосфат, а не никотина- мидмононуклеотид (NMN).
Терминация.
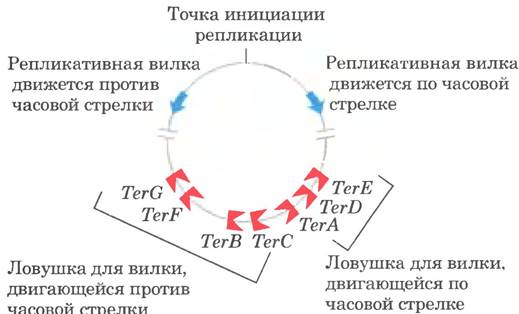
Наконец, две репликативные вилки кольцевой хромосомы Е. coli встречаются в точке терминации, содержащей множество копий последовательности из 20 п. н« называемой Теr (от лат. terminus — конец) (рис. 25-18). Последовательности Теr расположены на хромосоме таким образом, что создают что-то вроде ловушки, в которую репликативная вилка может войти, но не может из нее выйти. Теr-последовательности служат участками связывания белка Tus (от англ. terminus utilization substance). Комплекс Tus Ter может задержать репликативную вилку, движущуюся только в одном направлении. В каждом цикле репликации действует только один комплекс Tus- Теr — первый из тех, с которым столкнется первая вилка. Учитывая, что движущиеся в противоположных направлениях репликативные вилки обычно останавливаются при встрече, может показаться, что Теr-последовательности - необязательные элементы. Однако они могут предотвратить избыточную репликацию на одной из репликативных вилок, в случае если другая задерживается или останавливается при столкновении с повреждением ДНК или каким-либо другим препятствием.
Рис. 25-18. Терминация репликации хромосомы Е. coli. Последовательности Теr (от ТеrА до TerF) располагаются на хромосоме в виде двух кластеров с противоположной ориентацией.
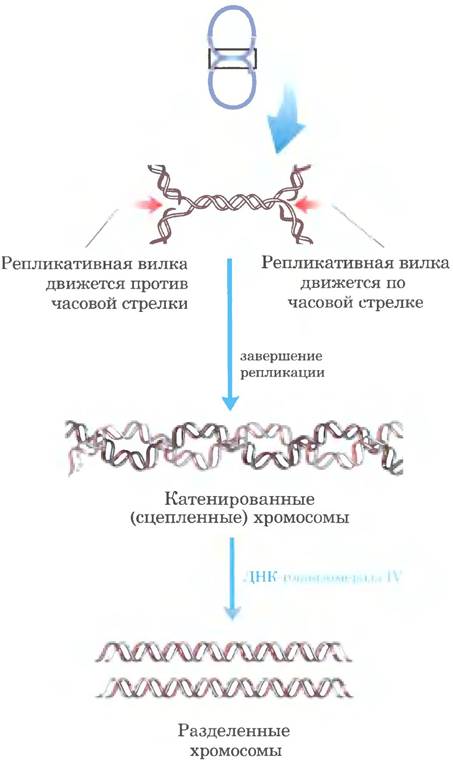
Таким образом, когда одна из репликативных вилок встречает функциональный комплекс Tus-Ter, она останавливается; а другая вилка останавливается, когда встречается с первой (задержанной) вилкой. Затем реплицируются последние несколько сотен пар оснований ДНК между этими крупными белковыми комплексами (по неизвестному механизму), достраивая две топологически сцепленные (катенированные) кольцевые хромосомы (рис. 25-19). Кольцевые ДНК, связанные таким образом, называются катенанами. Функцию разделения катенированных колец у Е. coli выполняет топоизомераза IV (топоизомераза II типа). Затем при клеточном делении разделенные хромосомы расходятся по дочерним клеткам. Заключительная фаза репликации других кольцевых хромосом, включая многие из ДНК-содержащих вирусов, инфицирующих эукариотические клетки, происходит по аналогичной схеме.
Рис. 25-19. Роль топоизомераз в терминации репликации. В результате репликации ДНК на двух противоположно направленных репликативных вилках образуются полноценные хромосомы, соединенные как катанены (топологически соединенные кольца). Кольца не связаны ковалентной связью, но поскольку они переплетены и каждое из них замкнуто ковалентно, их нельзя разделить без помощи топоизомераз. У Е. coli основную роль в разделении катенированных хромосом играет топоизомераза типа II (ДНК-топоизомераза IV), которая вносит кратковременный разрыв в обе цепи ДНК в одной хромосоме, позволяя другой хромосоме пройти сквозь разрыв.
Репликация в эукариотических клетках происходит по похожей схеме, но сложнее
Молекулы ДНК в эукариотических клетках значительно крупнее, чем в бактериях, и образуют сложные нуклеопротеиновые структуры (хроматин; разд. 24.3). Основная схема репликации ДНК у эукариот такая же, как у бактерий, и многие белковые комплексы похожи по функциям и структуре. Однако репликация у эукариот регулируется и координируется в соответствии с клеточным циклом, что усложняет процесс.
Участки начала репликации достаточно хорошо охарактеризованы у некоторых низших эукариот, но значительно хуже у высших эукариот. У позвоночных для инициации репликации могут использоваться разные А = Т-богатые последовательности, причем участки начала репликации могут меняться при каждом клеточном делении. Репликация дрожжей Saccharomyces cerevisiae начинается в определенной области, называемой автономно реплицирующейся последовательностью (АРП) или репликатором. Дрожжевые репликаторы имеют длину примерно 150 п. н. и содержат несколько важных консервативных последовательностей. В 16 хромосомах гаплоидного генома дрожжей рассредоточено около 400 репликаторов.
Механизмы регуляции обеспечивают однократную репликацию всей клеточной ДНК в каждом клеточном цикле. Важную роль в этой регуляции играют белки, называемые циклинами, и циклин-зависимые киназы (CDK), с которыми они образуют комплексы (с. 660 в т. 1). Циклины быстро разрушаются в результате убиквитин- зависимого протеолиза в конце М-фазы (фазы митоза), а отсутствие циклинов способствует организации пререпликативных комплексов (рrе- RC) в участках инициации репликации. В быстро растущих клетках комплекс pre-RC образуется в конце М-фазы. В медленно растущих клетках этот комплекс не образуется до конца фазы G1. Формирование комплексов pre-RC позволяет клетке начать репликацию (этот этап иногда называют лицензированием).
Как и у бактерий, ключевой момент в инициации репликации у всех эукариот — присоединение репликативной хеликазы, гетерогексамерного белкового комплекса МСМ (от англ. minichromosome maintenance; субъединицы от МСМ2 до МСМ7). Кольцевая хеликаза, состоящая из субъединиц МСМ2-7, функционирует подобно бактериальной хеликазе DnaB и связывается с ДНК при помощи другого комплекса из шести белков, называемого комплексом распознавания точки инициации репликации (ORC; от англ. origin recognition complex; рис. 25-20). Комплекс ORC состоит из пяти ААА+ АТРазных доменов и по своим функциям напоминает бактериальный комплекс DnaA. Кроме того, для присоединения комплекса МСМ2-7 необходимы два белка — CDC6 (от англ. cell division cycle) и CDT1 (от англ. CDC 10-dependent transcript 1), причем дрожжевой белок CDC6 представляет собой еще одну ААА+ АТРазу.
Рис. 25-20. Сборка пререпликативного комплекса в точке начала репликации у эукариот. В участке инициации репликации связываются белки ORC, CDC6 и CDT1. Эти белки, многие из которых являются ААА+ АТРазами, способствуют присоединению репликационной хеликазы МСМ2-7 в реакции, аналогичной реакции присоединения бактериальной хеликазы DnaB с помощью белка DnaC. В результате связывания хеликазного комплекса МСМ с ДНК образуется пререпликативный комплекс pre-RC, и именно эта стадия оказывается ключевой для инициации репликации.
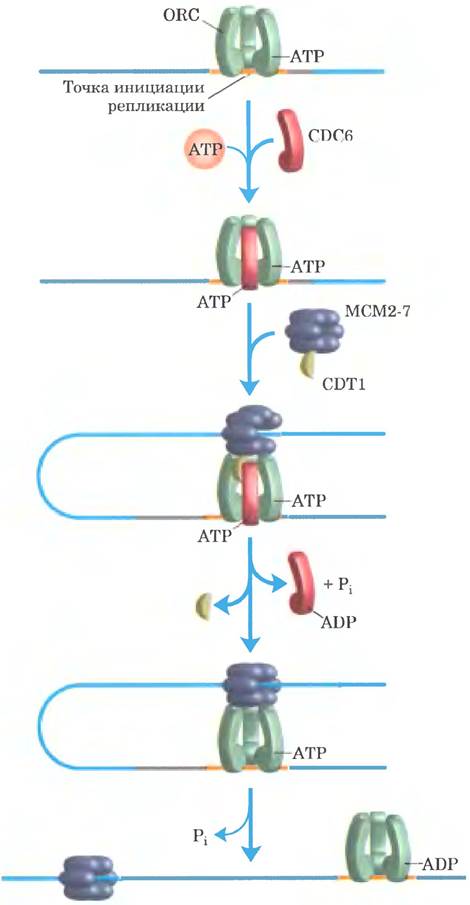
Для осуществления репликации в S-фазе происходит синтез и активация комплексов циклин-CDK (таких, как комплекс циклин Е- CDK2; см. рис. 12-45 в т. 1) и CDC7-DBF4. Оба типа комплексов помогают активизировать репликацию путем связывания и фосфорилирования некоторых белков пререпликативных комплексов. Другие циклины и CDK ингибируют образование дополнительных pre-RC комплексов после начала репликации. Например, CDK2 связывается с циклином А, когда уровень циклина Е снижается в ходе S-фазы, что ингибирует активность CDK2 и предотвращает образование дополнительных pre-RC-комплексов.
Скорость репликативной вилки у эукариот (около 50 нуклеотидов в секунду) примерно в 20 раз меньше, чем в Е. coli. Если бы репликация хромосомы человека начиналась в единственной точке инициации репликации, при такой скорости процесса репликация средней хромосомы продолжалась бы более 500 ч. На самом деле репликация хромосом человека происходит в двух направлениях и начинается во многих точках, удаленных друг от друга на расстояние от 30 до 300 т. п. н. Хромосомы эукариот почти всегда намного длиннее, чем хромосомы бактерий, поэтому наличие многих точек инициации репликации, вероятно, общее свойство клеток эукариот.
Как и у бактерий, у эукариот есть несколько типов ДНК-полимераз. Некоторые из них, возможно, выполняют особые функции, например, репликацию митохондриальной ДНК. В репликации ядерных хромосом участвует ДНК-полимераза а в комплексе с ДНК-полимеразой δ. Субъединичная ДНК-полимераза α имеет похожую структуру и свойства во всех эукариотических клетках. Одна из ее субъединиц выполняет функцию праймазы, а самая крупная субъединица (Мr ≈ 180 000) обладает полимеразной активностью. Однако данная полимераза не имеет корректирующей 3' —> 5'-экзонуклеазной активности и не может обеспечить высокой точности репликации. Предполагается, что ДНК-полимераза α нужна только для синтеза коротких праймеров (либо РНК, либо ДНК) для фрагментов Оказаки на отстающей цепи. Удлинение этих праймеров осуществляет мультисубъединичная ДНК -полимераза δ. Этот фермент связывается с белком, называемым ядерным антигеном пролиферирующих клеток (PCNA — от англ. proliferating cell nuclear antigen; Mr = 29 000), и стимулируется им; этот белок в большом количестве содержится в ядрах делящихся клеток. Трехмерная структура PCNA удивительным образом напоминает структуру β-субъединицы ДНК-полимеразы III Е. соli (рис. 25-10, б), хотя первичные последовательности этих белков не имеют выраженной гомологии. Функционально PCNA представляет собой аналог β-субъединиц; он формирует кольцевой зажим, который существенно увеличивает процессивность полимеразы. ДНК-полимераза δ обладает корректирующей 3' —> 5'-экзонуклеазной активностью и, предположительно, обеспечивает синтез как лидирующей, так и отстающей цепи в комплексе, напоминающем димерную бактериальную ДНК-полимеразу III.
Еще одна полимераза, ДНК-полимераза ε, в некоторых ситуациях заменяет ДНК-полимеразу δ, например, при репарации ДНК. ДНК-полимераза е также может действовать в репликативной вилке, возможно, аналогично бактериальной ДНК-полимеразе I, удаляя праймеры фрагментов Оказаки на отстающей цепи.
В репликации ДНК у эукариот задействованы еще два белковых комплекса. Белок RPA (от англ. replication protein А) связывает одноцепочечную ДНК, и его функция аналогична функции белка SSB в клетках Е. coli Белок RFC (от англ/ replication factor С) представляет собой погрузчик зажима для PCNA, который облегчает сборку активных репликационных комплексов. Аминокислотные последовательности субъединиц комплекса RFC имеют значительное сходство с последовательностями субъединиц бактериального y-комплекса.
Для терминации репликации линейных эукариотических хромосом на концах каждой хромосомы синтезируются специальные структуры, называемые теломерами (см. гл. 26).
Вирусные ДНК-полимеразы являются мишенями для противовирусной терапии
Многие ДНК-co держащие вирусы кодируют собственные ДНК-полимеразы, и некоторые из них становятся мишенями для лекарственных средств. Например, ДНК-полимераза вируса простого герпеса ингибируется ацикловиром, разработанным Гертрудой Элайон (см. с. 558 в т. 2). Ацикловир состоит из гуанина, к которому присоединено неполное кольцо рибозы.
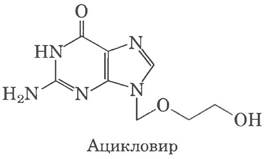
Ацикловир фосфорилируется вирусной тимидинкиназой. Сродство ацикловира к вирусному ферменту в 200 раз выше сродства к клеточной тимидинкиназе. В результате фосфорилирование происходит преимущественно в клетках, инфицированных вирусом. Клеточные киназы превращают образовавшийся ацикло-GMP в ацикло-GTP, который одновременно является и ингибитором, и субстратом ДНК-полимераз. Ацикло-GTP сильнее ингибирует ДНК-полимеразу вируса герпеса, чем клеточные ДНК-полимеразы. Поскольку ацикло-GTP не имеет 3'-гидроксильной группы, его включение в цепь ДНК воспринимается как сигнал терминации. Таким образом, репликация вируса ингибируется на нескольких стадиях. ■
Краткое содержание раздела 25.1 Репликация ДНК
■ Репликация ДНК происходит с очень высокой точностью и в определенной фазе клеточного цикла. Репликация полуконсервативна, каждая цепь выступает матрицей для новой дочерней цепи. Репликация протекает в три стадии: инициация, элонгация и терминация. В бактериях реакция начинается в точке инициации репликации и обычно происходит в двух направлениях.
■ ДНК синтезируется ДНК-полимеразами в направлении 5' —> 3'. В репликативной вилке лидирующая цепь синтезируется непрерывно по ходу движения репликативной вилки; отстающая цепь синтезируется прерывисто в виде фрагментов Оказаки, которые затем сшиваются.
■ Точность репликации ДНК достигается (1) путем выбора оснований полимеразой, (2) с помощью корректирующей 3' —> 5'-экзонуклеазной активности, которой обладает большинство ДНК-полимераз, и (3) с помощью специфических систем репарации, исправляющими ошибки, оставшиеся после репликации.
■ Большинство клеток имеет несколько ДНК-полимераз. У Е. coli основной фермент репликации — ДНК-полимераза III. ДНК-полимераза I выполняет особые функции в ходе репликации, рекомбинации и репарации.
■ На стадиях инициации, элонгации и терминации репликации ДНК работает множество ферментов и белковых факторов, многие из них принадлежат к семейству ААА+ АТРаз.
■ Белки бактерий, участвующие в репликации, организованы в крупные комплексы, внутри которых матрица ДНК протягивается через две реплисомы, связанные с плазматической мембраной.