Молекулярная биология клетки - Том 2 - Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К., Уотсон Дж. 1993
Контроль генной экспрессии
Организация и эволюция ядерного генома
В геномах ныне живущих организмов записана значительная часть их эволюционной истории. Некоторые ее страницы могут быть расшифрованы при изучении последовательности ДНК этих организмов. Методы секвенирования ДНК, широко распространенные в настоящее время, дают возможность анализировать большое количество молекул ДНК и судить о том, как за десятки миллионов лет возникли гены, кодирующие определенные белки. Изучение случайных изменений, происходящих в хромосомах сейчас, проливает дополнительный свет на механизмы, ответственные за эволюционные изменения в прошлом. В данном разделе представлены некоторые молекулярно-генетические подходы, направленные на изучение организации и эволюции ядерного генома высших эукариот.
10-32
10-34
10.5.1. Точковые мутации обусловливают небольшие изменения генома, а его перестройка или увеличение осуществляются в ходе генетической рекомбинации [54]
Последовательность нуклеотидов в ДНК должна точно реплицироваться и сохраняться. В гл. 5 обсуждались сложные механизмы, позволяющие ДНК наследоваться с необычайной точностью: за каждые 200000 лет случайно меняется лишь одна нуклеотидная пара из тысячи (см. разд. 5.2). И даже при такой скорости мутирования в популяции, состоящей из 10000 особей, каждая возможная нуклеотидная замена будет «испробована» около пятидесяти раз за миллион лет. Если какой-либо вариант последовательности обладает преимуществом, он быстро размножится благодаря естественному отбору. Следовательно, можно ожидать, что у любого вида функция большинства генов будет оптимизирована в отношении вариаций, возникающих вследствие точковых мутаций.
Точковые мутации служат для тонкой «подстройки» генома, но долговременный эволюционный процесс должен быть связан с более радикальными генетическими изменениями. Эту функцию выполняет генетическая рекомбинация; с ее помощью геном может увеличиваться или уменьшаться (при дупликации или делеции), а его части могут перемещаться из одной области в другую, образуя новые комбинации. Составляющие части генов (их экзоны и регуляторные элементы) могут перемешиваться, давая начало новым белкам, обладающим совершенно новыми функциями. Кроме того, если какой-либо ген представлен в геноме двумя копиями, одна из них может подвергнуться мутации, что приведет к дивергенции копий и их специализации для едва различающихся функций. Таким путем геном как целое постепенно усложняется и совершенствуется. Например, у млекопитающих почти каждый ген существует в нескольких вариантах: разные гены актина - для различных типов сократительных клеток, разные гены родопсина - для восприятия различных цветов, разные гены коллагена - для различных типов соединительных тканей и так далее. Экспрессия каждого гена регулируется строго и специфически. Изучение последовательностей ДНК показывает, что многие гены, даже значительно отличающиеся друг от друга, могут иметь родственные модульные области. Так например, определенная часть генов родопсина имеет общего предшественника с рядом генов, кодирующих некоторые гормоны и рецепторы (см. разд. 12.3.13); эта общая последовательность, вероятно, присутствует и в других белках (см. разд. 3.3.8).
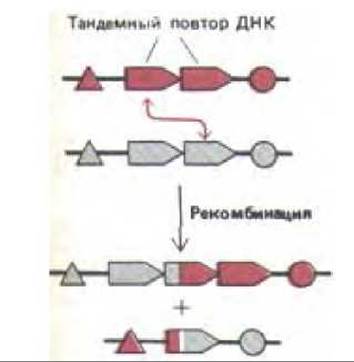
Рис. 10-63. Семейство тандемно повторенных генов теряет и восстанавливает свои копии вследствие кроссинговера между сестринскими хромосомами, несущими эти гены. Это происходит довольно часто, так как длинные участки гомологичных последовательностей ДНК являются хорошим субстратом для общей генетической рекомбинации.
Основой для возникновения подобных семейств генов и генных сегментов служит генетическая рекомбинация. Выше обсуждались молекулярные механизмы общей и сайт-специфической рекомбинации. В данном разделе будут рассмотрены некоторые результаты воздействия рекомбинации на геном.
10-33
10-34
10-35
10.5.2. Тандемно повторяющиеся последовательности ДНК стремятся остаться неизмененными [55]
Дупликации генов обычно объясняют редкими событиями, которые катализируются некоторыми рекомбинационными ферментами. Однако у высших эукариот имеется эффективная ферментативная система, которая соединяет концы разорванной молекулы ДНК. Таким образом, дупликации (а также инверсии, делеции и транслокации сегментов ДНК) могут возникать у этих организмов вследствие ошибочного воссоединения фрагментов хромосомы, которая по каким-то причинам оказалась разорванной. Если дуплицированные последовательности соединяются «голова к хвосту», то говорят о тандемных повторах. Появление одного тандемного повтора легко может привести к возникновению их длинной серии в результате неравного кроссинговера между двумя сестринскими хромосомами, поскольку длинные участки спаривающихся последовательностей представляют собой идеальный субстрат для обычной рекомбинации (рис. 10-63). Дупликация ДНК и следующий за ней неравный кроссинговер лежат в основе амплификации ДНК, процесса, который, как выяснилось, способствует возникновению раковых клеток (см. рис. 21-26). В ходе неравного кроссинговера число тандемно повторяющихся генов может как увеличиваться, так и уменьшаться (см. рис, 10-63). Большое количество повторяющихся генов будет поддерживаться естественным отбором лишь в том случае, если существование дополнительных копий окажется выгодным для организма. Как отмечалось выше, у позвоночных тандемный повтор кодирует большой предшественник рибосомной РНК, что необходимо для обеспечения потребности растущих клеток в новых рибосомах (см. разд. 9.4.16). Кластеры тандемно повторяющихся генов кодируют у позвоночных и другие структурные РНК, включая 5S-pPHK, U1- и U2-мяРНК. Тандемные повторы характерны и для гистоновых генов, на которых синтезируется большое количество белка, требующегося в каждой S-фазе.
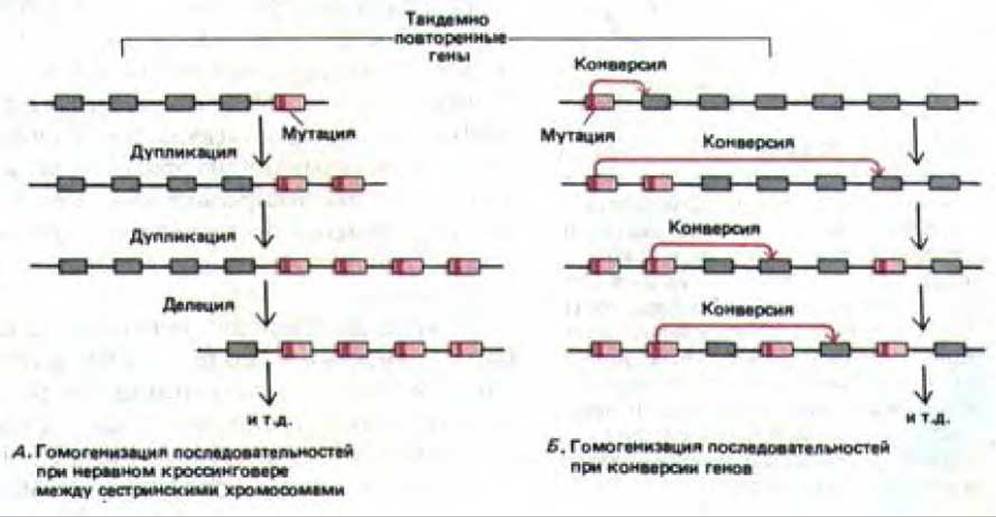
Рис. 10-64. Два типа событий, позволяющих сохранить последовательности ДНК в тандемном расположении и очень похожими друг на друга. А. Постоянное увеличение и уменьшение числа копий гена в тандеме при неравном кроссинговере (см. рис. 10-63) приводит к гомогенизации всех последовательностей генов, входящих в состав кластера. Б. При конверсии генов одна копия действует как матрица, которая передает либо все. либо часть последовательностей своей ДНК другой копии гена. У высших эукариот эти процессы, по-видимому, присущи генам, расположенным рядом друг с другом на хромосоме. У низших эукариот, например у грибов, конверсия генов у которых изучена гораздо лучше, этот процесс, как оказалось, не лимитирован лишь соседними генами.
Можно ожидать, что в ходе эволюции последовательности тандемно расположенных генов, а также нетранскрибируемой ДНК спенсеров, расположенных между ними, дивергируют за счет случайных мутаций. изменяющих одну или несколько копий гена. Однако на самом деле последовательности тандемно повторенных генов и их спейсерная ДНК обычно почти идентичны. Полагают, что к этому причастны два механизма: во-первых, неравный кроссинговер, приводящий к последовательному расширению и сокращению областей, содержащих тандемно повторяющиеся последовательности (анализ компьютерной модели такого кроссинговера показывает, что при этом последовательности имеют тенденцию оставаться прежними, рис. 10-64, А); во-вторых, конверсия генов (показано, что она может обусловливать гомогенизацию родственных последовательностей ДНК, рис. 10-64, Б).
10.5.3. На примере семейства глобиновых генов можно проследить, как случайные дупликации ДНК способствуют эволюции организмов [56]
Дупликации ДНК имеют очень большое значение для эволюции новых белков. Чтобы убедиться в этом, обратимся к семейству глобиновых генов, поскольку его история изучена особенно хорошо. Явные гомологии в аминокислотной последовательности и в структуре современных глобиновых генов указывают на их происхождение от общего предка, несмотря на то, что некоторые члены этого семейства теперь расположены в геноме млекопитающих в совершенно разных местах.
Анализируя формы гемоглобина в организмах, стоящих на разных ступенях филогенетической лестницы, можно восстановить некоторые события, приведшие к возникновению разнообразных типов этого белка. Появление гемоглобиноподобных молекул в ходе эволюции, по- видимому, способствовало увеличению размеров многоклеточных животных. Крупным животным для поддержания должного уровня кислорода в тканях уже недостаточно простой диффузии. В результате, гемоглобиновые молекулы обнаруживаются у всех позвоночных и многих беспозвоночных. Самая примитивная молекула, переносящая кислород, представляет собой глобиновую полипептидную цепь размером около 150 аминокислот. Она обнаруживается у многих морских червей, насекомых и примитивных рыб. Молекула гемоглобина у высших позвоночных устроена более сложно: в ее состав входит два типа глобиновых цепей. По-видимому, около 500 млн лет назад в ходе эволюции высших рыб произошла серия мутаций и дупликации соответствующего гена. В результате этих событий вначале образовалось два слегка отличающихся друг от друга гена, кодирующих цепи а- и ß-глобинов в геноме каждой особи. У современных высших позвоночных каждая молекула гемоглобина представляет собой комплекс, состоящий из двух а- и двух ß-цепей. (рис. 10-65). Такая структура функционирует гораздо более эффективно, чем молекула гемоглобина, содержащая одну цепь. Четыре кислород-связывающих сайта в молекуле a2ß2 взаимодействуют друг с другом. Это взаимодействие приводит к кооперативному аллостерическому изменению в молекуле при связывании и освобождении кислорода, позволяющему доставлять в ткани гораздо большие порции кислорода.
В ходе дальнейшей эволюции млекопитающих мутации и дупликации, по-видимому, подвергся ген ß-цепи, вследствие чего возник второй тип гемоглобина, синтезируемый только в эмбрионе. Образовавшаяся молекула гемоглобина обладает повышенным сродством к кислороду по сравнению с гемоглобином взрослой особи, и, таким образом, способствует переносу кислорода от матери к плоду. Ген, кодирующий новую, похожую на ß-цепь, молекулу гемоглобина, вновь подвергся последовательным мутациям и дупликациям, в ходе которых возникли два новых гена ε и у. Цепь є синтезируется на более ранних стадиях развития (с образованием а2ε2), чем у-цепь эмбриона, образующая форму а2y2 (см. рис. 10-39, Б). Дупликация гена ß-цепи взрослых, происшедшая еще позже в ходе эволюции приматов, привела к образованию гена 5-глобина и соответственно минорной формы глобина (а2δ2), обнаруживаемой только у взрослых приматов (рис. 10-66). Каждый из этих дуплицировавшихся генов впоследствии был модифицирован за счет точковых мутаций, воздействующих на свойства конечной молекулы гемоглобина, а также в результате изменений в регуляторных областях, определяющих выбор времени и уровень экспрессии данного гена (см. рис. 10-73).
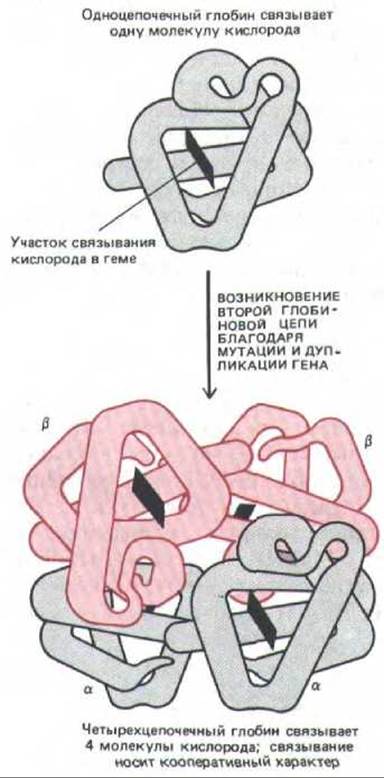
Рис. 10-65. Пространственная структура одноцепочечного и четырехцепочечного глобинов. Изображенный здесь четырехцепочечный гемоглобин представляет собой комплекс, состоящий из двух а- и двух ß-глобиновых цепей. Глобин, состоящий из одной цепи, у некоторых примитивных позвоночных образует димер, который диссоциирует при связывании кислорода и представляет собой промежуточную ступень в эволюции глобина, содержащего четыре цепи.
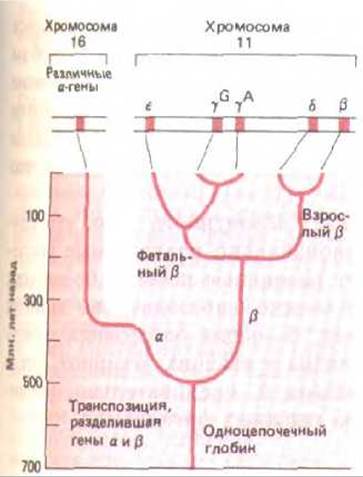
Рис. 10-66. Схема эволюции цепей глобина на примере семейства ß-подобных глобиновых генов (см. рис. 10-39). Относительно недавно возникшие дупликации гена у-цепи, привели к образованию уG- и уА-цепей, которые являются 3-подобными и обладают идентичными функциями.
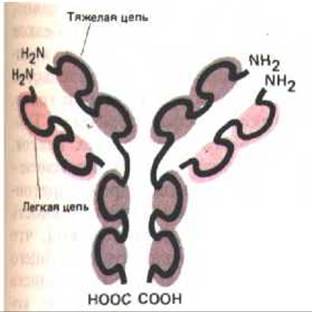
Рис. 10-67. Структура молекулы антитела (иммуноглобулинов). Эта молекула состоит из двух идентичных тяжелых цепей и двух идентичных легких цепей (выделены цветом). Каждая тяжелая цепь содержит четыре сходных ковалентно связанных домена. Каждая легкая цепь имеет в своем составе два таких домена. Каждый домен кодируется отдельным экзоном; вероятно, все экзоны возникли в результате дупликации одного исходного экзона.
Конечный результат процесса дупликации генов, приведшего к дивергенции глобиновых цепей, хорошо виден при рассмотрении генов, возникших из исходного ß-гена и расположенных в виде серии гомологичных последовательностей ДНК внутри сегмента ДНК размером 50000 нуклеотидных пар (см. рис. 10-39, А). У человека кластер а-глобиновых генов находится на другой хромосоме. На основании того, что у птиц и млекопитающих кластеры а- и ß-глобиновых генов обнаруживаются в разных хромосомах, а у лягушки Xenopus они лежат рядом, считается, что два гена разъединились в результате транслокации примерно 300 млн лет назад (рис. 10-66). Подобные транслокации, вероятно, способствуют стабилизации дуплицированных генов, обладающих различными функциями, поскольку предохраняют их от гомогенизации, которой часто подвергаются близлежащие гены со сходной последовательностью (см. рис. 10-64).
Существует несколько дуплицированных последовательностей глобиновой ДНК, входящей в состав кластеров а- и ß-глобиновых генов, которые не являются активными. Это пример псевдогенов, которые имеют высокую степень гомологии с активными генами, но неактивны вследствие мутаций, препятствующих их экспрессии. Существование подобных псевдогенов не должно вызывать удивления, ведь не все дупликации ДНК могут приводить к возникновению новых активных генов, между тем неактивные последовательности не удаляются из генома немедленно.
Сравнивая последовательности ДНК многих семейств генов у животных, стоящих на разных ступенях филогенеза, можно проследить значительную часть истории нашей эволюции (см. рис. 4-62).
10.5.4. Гены, кодирующие новые белки, могут образовываться при рекомбинации экзонов [54]
Роль дупликации ДНК в эволюции не ограничивается их участием в образовании больших генных семейств. Дупликации могут иметь значение и для возникновения новых одиночных генов. Белки, кодируемые такими генами, можно узнать по присутствию в них повторяющихся сходных белковых доменов, которые последовательно ковалентно связаны друг с другом. Например, иммуноглобулины (рис. 10-67), альбумины, а также большинство фибриллярных белков (таких, как спектрины и коллагены) кодируются генами, возникшими в результате многократных дупликации исходной последовательности ДНК.
У генов, возникших таким путем, каждый экзон часто кодирует отдельную субъединицу или домен в белке (см. разд. 3.3.4). Организация кодирующих последовательностей ДНК в виде серии таких экзонов, разделенных длинными нитронами, в значительной мере упростила эволюцию новых белков. Например, дупликации, необходимые для образования отдельного гена, кодирующего белок с повторяющимися доменами, могут возникать при разрыве и воссоединении ДНК в любом месте длинных нитронов, окружающих экзон. Без нитронов в исходном гене было бы лишь несколько сайтов, рекомбинация по которым могла бы привести к дупликации домена. Увеличивая число возможных сайтов для дупликации, интроны значительно повышают вероятность того, что дупликация окажется полезной.
Наличие нитронов намного увеличивает вероятность того, что случайная рекомбинация соединит две первоначально разделенные последовательности ДНК, которые кодируют различные домены белка (см, рис. 10-71). Результаты таких событий можно наблюдать во многих современных белках (см. рис. 3-38). Итак, большие расстояния между экзонами, кодирующими отдельные домены у высших эукариот, ускоряют процесс возникновения новых белков и, следовательно, увеличивают эффективность эволюции весьма сложных организмов.
10.5.5. Вероятно, большинство белков кодируются генами, состоящими из многих небольших экзонов [57]
Открытие в 1977 г. прерывистости генов эукариот оказалось совершенно неожиданным. Все исследованные до этого гены были бактериального происхождения и не содержали нитронов. У бактерий, как известно, отсутствуют ядро и внутренние мембраны, их геном меньше, чем геном эукариот, и традиционно считалось, что бактерия напоминает ту древнюю простую клетку, из которой произошла клетка эукариотическая. Неудивительно поэтому, что многие биологи вначале воспринимали интроны как причудливую позднейшую эволюционную добавку. Однако в настоящее время все больше утверждается точка зрения, согласно которой прерывистые гены имеют весьма древнее происхождение, а бактерии потеряли свои интроны лишь после того, как возникла большая часть их белков.
Мысль о том, что интроны появились в ходе эволюции очень давно соответствует современному представлению о происхождении белков методом проб и ошибок при рекомбинации отдельных экзонов, кодирующих различающиеся белковые домены. Более того, доказательства древнего происхождения нитронов были получены при изучении генов, кодирующих распространенный фермент триозофосфатизомеразу. Триозофосфатизомераза играет важную роль в метаболизме всех клеток, катализируя центральное событие при гликолизе и глюконеогенезевзаимопревращение глицералальдегида-3-фосфата и дигидроксиацетон-фосфата (см. рис. 2-38). Сравнивая аминокислотную последовательность этого фермента у различных организмов, можно сделать вывод, что фермент возник еще до дивергенции прокариот и эукариот от общего предка, поскольку 46% аминокислотной последовательности у человека и бактерии идентичны. У позвоночных (курицы и человека) ген, кодирующий этот фермент, содержит шесть нитронов, причем пять из них присутствуют точно в том же месте у кукурузы. Из этого следует, что эти пять нитронов существовали в гене до того, как растения и животные дивергировали в ходе эволюции эукариот, что, как установлено, произошло 109 лет назад (рис. 10-68).
Мелкие одноклеточные организмы находятся под сильным давлением отбора, что заставило их воспроизводиться путем деления клеток с максимальной скоростью, какую только позволяет содержание питательных веществ в окружающей среде. В связи с этим, они вынуждены свести к минимуму содержание ненужной ДНК, которую надо синтезировать в каждом цикле клеточного деления. Для организмов большего размера, живущих благодаря хищничеству, и в целом для многоклеточных организмов, у которых скорости деления клеток определяются другими причинами, такое сильное давление отбора, ведущее к удалению избыточной ДНК из генома, отсутствует. Скорее всего именно эти обстоятельства объясняют, почему бактерии должны были потерять свои интроны, тогда как эукариоты их сохранили. Подобное объяснение соответствует также другим данным, полученным при изучении триозофосфатизомеразы: в то время как многоклеточный гриб Aspergillus содержит пять нитронов в гене, кодирующем этот фермент, его одноклеточный родич, дрожжи Saccharomyces, не содержит их вообще.
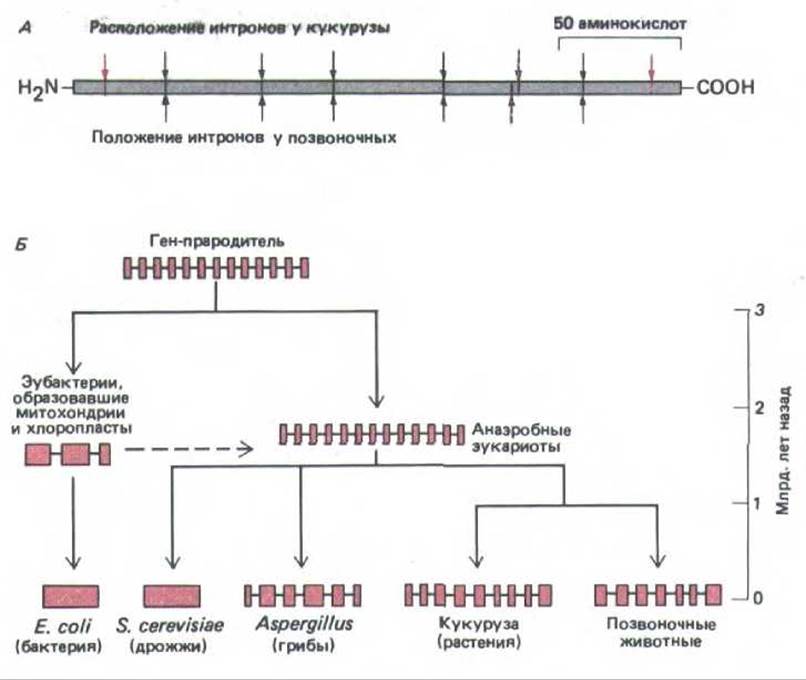
Рис. 10-68. Эволюция прерывистых генов с древнейших времен. А. Структура гена триозофосфатизомеразы у растений и животных. Одинаковые положения нитронов у кукурузы (зерна) и позвоночных отмечены черными стрелками, а отличающиеся положения выделены красными стрелками. Так как считается, что растения и животные возникли от общего предка около миллиарда лет назад, общие интроны должны иметь очень древнее происхождение. Б, Гипотетическая схема возникновения определенного гена. Последовательности экзонов выделены цветом, а последовательности интрона обозначены черным. Приведенный здесь ген кодирует белок, необходимый для всех клеток. Подобно триозофосфатизомеразе, этот белок, по-видимому, сформировал свою окончательную трехмерную структуру перед тем, как от общего предка отделились бактерии, архебактерии и эукариоты. Этот общий предок обозначен на рисунке как «ген-прародитель». Пунктиром указано примерное время прохождения эндосимбиотических процессов, которые привели к возникновению митохондрий и хлоропластов. (А-nо W. Gilbert, M. Marchionni and G.Mc. Knight, Cell 46: 151-154, 1987.)
Каков же механизм потери нитронов? Возможно, интроны терялись при постепенных случайных делециях коротких сегментов ДНК, но более вероятно, что эукариотические клетки (а возможно, также и предки бактерий) имеют механизм точной и селективной делеции всего интрона из своих геномов. Например, в клетках большинства позвоночных содержится лишь один ген инсулина с двумя нитронами, но у крыс no-соседству имеется еще один инсулиновый ген, в составе которого всего один интрон. Очевидно, второй ген возник относительно недавно в результате дупликации и затем потерял один из своих нитронов. Так как при потере интрона необходимо точное воссоединение кодирующих последовательностей ДНК, считается, что второй ген возник в результате редкого события - включения в геном ДНК-копии мРНК соответствующего гена, откуда интроны были точно удалены. Подобные копии, не содержащие нитронов, могут появляться благодаря активности обратных транскриптаз. Считают, что ферменты рекомбинации дают возможность таким копиям спариться с исходной последовательностью, которая затем «корректируется» по матрице, лишенной нитронов, в ходе событий, напоминающих конверсию гена.
Обратные транскриптазы синтезируются в клетках транспозируемыми элементами (см. табл. 10-3) и всеми ретровирусами. Образование ДНК-копий частей генома при обратной транскрипции, по-видимому, тоже внесло свой вклад в эволюцию геномов высших организмов.
10.5.6. Основная фракция ДНК высших эукариот состоит из повторяющихся некодирующих последовательностей нуклеотидов [58]
Геномы эукариот содержат не только интроны, но также и большое число копий ДНК, которая не кодирует белок и представляется ненужной. Присутствие таких повторяющихся последовательностей ДНК у высших эукариот впервые было обнаружено методом гибридизации, позволяющим оценить число копий гена (см. разд. 4.6.7). При использовании этого метода геном механически нарезается на короткие двухцепочечные фрагменты длиной около 1000 нуклеотидных пар; затем фрагменты подвергают денатурации и получают одноцепочечную ДНК. Скорость, с которой одноцепочечные ДНК гибридизуются в смеси, зависит от комплементарности фрагментов. Для большей части фрагментов реакция протекает очень медленно. Например, гаплоидный геном клетки млекопитающего представлен примерно 6 млн различных фрагментов ДНК длиной 1000 нуклеотидов, и любой фрагмент, последовательность которого содержится лишь в одной копии, должен случайно столкнуться с 6 млн некомплементарных цепей, чтобы наш гомолога.
Анализ гибридизации ДНК из клеток человека показал, что примерно 70% одноцепочечных фрагментов гибридизуется очень медленно, т. е. так, как и следует ожидать в случае большого набора уникальных (неповторяющихся) последовательностей (полная гибридизация присходит в течение нескольких дней). Однако остальные 30% цепей ДНК гибридизуется гораздо быстрее. Эти цепи содержат последовательности, которые многократно повторены в геноме, и, следовательно, могут относительно быстро найти своего партнера. Большая часть такой ДНК не кодирует белки, приблизительно одну ее треть составляют тандемно повторяющиеся сателлитные последовательности, остальные две трети приходятся на рассеянную по геному повторяющуюся ДНК. Эти диспергированные повторы, по-видимому, произошли из транспозируемых элементов, размножившихся в нашем геноме и достигших исключительно высокой степени копийности.
10.5.7. Функция сателлитной ДНК неизвестна [59]
Большая часть быстро гибридизующихся цепей ДНК обычно состоит из очень длинных тандемных повторов одной короткой последовательности нуклеотидов (рис. 10-69). Повторяющаяся единица в подобной последовательности может быть представлена даже одним или двумя нуклеотидами, однако большинство повторов длиннее; у млекопитающих они обычно составлены из вариантов одной короткой последовательности, организованной в повтор размером в несколько сот нуклеотидов. Такие тандемные повторы простой последовательности называются сателлитной ДНК, поскольку первая обнаруженная ДНК такого типа имела необычный нуклеотидный состав, что давало возможность отделить ее от тотальной клеточной ДНК в виде минорного компонента (или «сателлита»). Обычно последовательности сателлитной ДНК не транскрибируются и чаще всего локализованы в гетерохроматине центромерных областей хромосом (см. разд. 10.3.8). У некоторых млекопитающих на долю сателлитной ДНК приходится 10% и более от всей ДНК (сателлитные последовательности могут даже занимать целое плечо хромосомы).
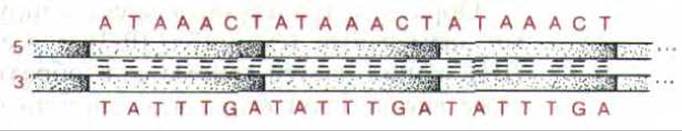
Рис. 10-69. Одна из последовательностей сателлитной ДНК, образованная серией повторяющихся блоков из семи нуклеотидов. Данная последовательность обнаружена в геноме дрозофилы.
Последовательности сателлитной ДНК способны быстро меняться; более того, в ходе эволюции произошел их сдвиг по хромосоме. Например, при сравнении двух гомологичных хромосом у любого человека некоторые последовательности сателлитной ДНК обнаруживаются у них в разных местах. Обычно у двух близкородственных видов последовательности сателлитной ДНК значительно отличаются, между тем последовательности ДНК в других областях генома характеризуются высокой консервативностью. До сих пор неизвестно, какие функции имеют последовательности сателлитной ДНК: все попытки доказать ее участие в спаривании хромосом или организации ядра заканчивались неудачей. Было выдвинуто предположение, что это «эгоистичная ДНК», которая «заботится» лишь о сохранении своих последовательностей в составе генома, но никак не способствует выживанию содержащих её клеток. Другие последовательности, которые обычно рассматривают как эгоистичную ДНК, - это транспозирующиеся элементы или транспозоны.
10-36
10.5.8. Эволюция геномов ускоряется транспозирующимися элементами по крайней мере трех типов [60]
Геномы обычно содержат много разнообразных транспозирующихся элементов, или транспозовов. Впервые эти элементы были обнаружены в геноме кукурузы; некоторые из них удалось охарактеризовать и даже определить их первичную структуру. Лучше всего изучены транспозоны у дрозофилы, где известно более 30 их типов. Длина этих транспозонов колеблется от 2000 до 10000 нуклеотидных пар; большинство из них присутствует в геноме в количестве 5-10 копий на диплоидную клетку. В настоящее время различают три больших класса транспозонов на основании особенностей организации их последовательностей (табл. 10-3). Некоторые элементы перемещаются в геноме в виде ДНК, но есть и такие, у которых этот процесс включает образование промежуточного продукта (в его роли выступает РНК). В любом случае транспозоны способны размножаться, вырезаться из каких-то сайтов и внедряться в другие; их поведение можно охарактеризовать как паразитическое.
Таблица 10-3. Три основных семейства транспозонов

Длина этих элементов варьирует от 2000 до 12 000 нуклеотидных пар. В состав каждого семейства входит много различных элементов, в таблице приведены лишь немногие из них.
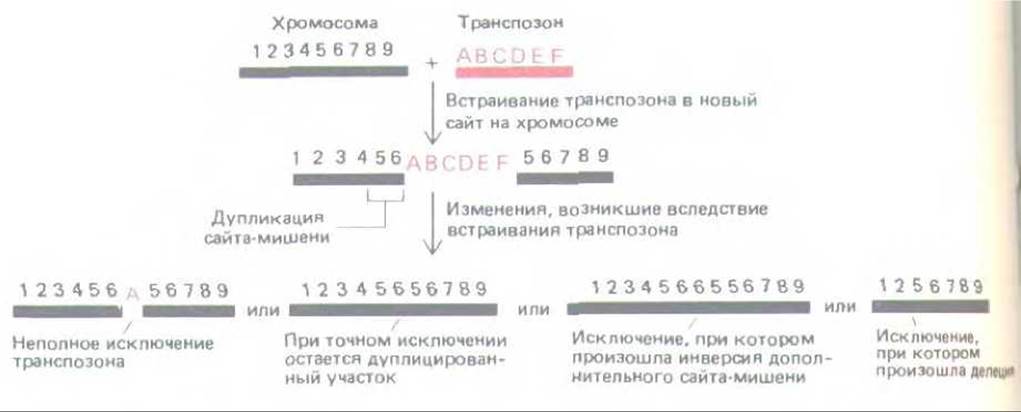
Рис. 10-70: Некоторые изменения в последовательностях ДНК хромосомы, возникающие вследствие перемещения транспозонов. При встраивании транспозона всегда образуется короткая дупликация хромосомной последовательности из 3-12 нуклеотидных пар. Ферменты сайтспецифической рекомбинации, кодируемые этим элементом, участвуют и в последующем исключении транспозона. При таком исключении последовательность хромосомной ДНК часто не восстанавливается, как это показано на четырех примерах.
На долю транспоэонов приходится по крайней мере 10% геномнов ДНК высших эукариот. Большинство этих элементов перемещается лишь изредка, но поскольку их количество в клетке велико, транспозиция оказывает значительное влияние на разнообразие видов. Например, больше половины спонтанных мутаций, изученных у дрозофилы, вызвана встраиванием транспозона внутрь мутировавшего гена или вблизи него.
Мутации могут возникать либо когда элемент встраивается в ген, либо когда он начинает перемещаться в какое-либо другое место. Все известные транспозоны приводят к появлению коротких дупликаций в сайте-мишени, что связано с механизмом их встраивания (см. рис. 5-67, Б). При вырезании транспозона из хромосомы обычно он оставляет на месте своего пребывания одну из копий, составляющих дупликацию (рис. 1070). Таким образом, перемещение транспозона сопровождается вставками и делециями в нуклеотидной последовательности.
Транспозоны вносят свой вклад в вариабельность генома и иными средствами. Если два транспозона, которые узнаются одним и тем же сайт-специфическим ферментом рекомбинации (транспозазоп), встраиваются в соседние сайты хромосомы, ДНК между ними может стать субстратом для транспозиции, осуществляемой с помощью транспозазы. Так как это весьма эффективный путь перемещения экзонов, справедливо утверждение, что транспозоны могут способствовать образованию новых генов (рис. 10-71).
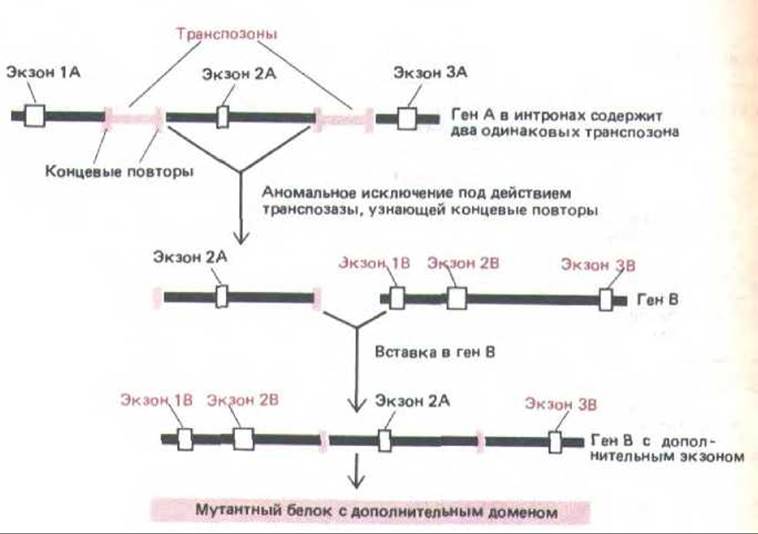
Рис. 10-71. Перемещение экзона, которое может происходить в результате встраивания транспозонов. Когда два транспозона, принадлежащие к одному и тому же типу (выделены цветом), оказываются по соседству друг с другом на хромосоме, то в транспозиции могут оказаться задействованными концы двух разных элементов (вместо двух концов одного и того же элемента); в результате хромосомная ДНК, заключенная между ними, переместится в новую область хромосомы. Так как по сравнению с экзонами интроны очень велики (см. рис. 9-7), изображенное на схеме встраивание нового экзона в ранее существовавший интрон - не такое уж невероятное событие.
10-36
10.5.9. Транспозоны могут влиять на регуляцию генов [61]
Перестройки последовательностей ДНК, вызываемые транспозонами, часто изменяют экспрессию близлежащих генов, что может привести к различным нарушениям в развитии животных или растений, например их пигментации (рис. 10-72). Большая часть таких изменений в регуляции генов, как правило, оказывается вредной для организма, но некоторые - могут оказаться и полезными.
Свойства мутаций, вызываемых транспозонами, необычны и позволяют отличить их от мутаций, возникших вследствие ошибок в репликации или репарации ДНК. Одно важное отличие состоит в том, что при перемещении транспозона вблизи гена часто оказываются новые последовательности, которые действуют как участки узнавания для сайт-специфических ДНК-связывающих белков, включая транспозазу и белки, регулирующие транскрипцию ДНК, входящей в состав транспозона. Таким образом, эти последовательности могут действовать как энхансеры и усиливать транскрипцию генов, расположенных от них на расстоянии тысяч нуклеотидных пар. Пример такого типа воздействия на экспрессию гена пигмента у кукурузы приведен на рис. 10-72. Аналогичный эффект может вносить свой вклад в возникновение раковых клеток: перенос регуляторных последовательностей в области, соседствующие с протоонкогеном, способен превратить его в онкоген.
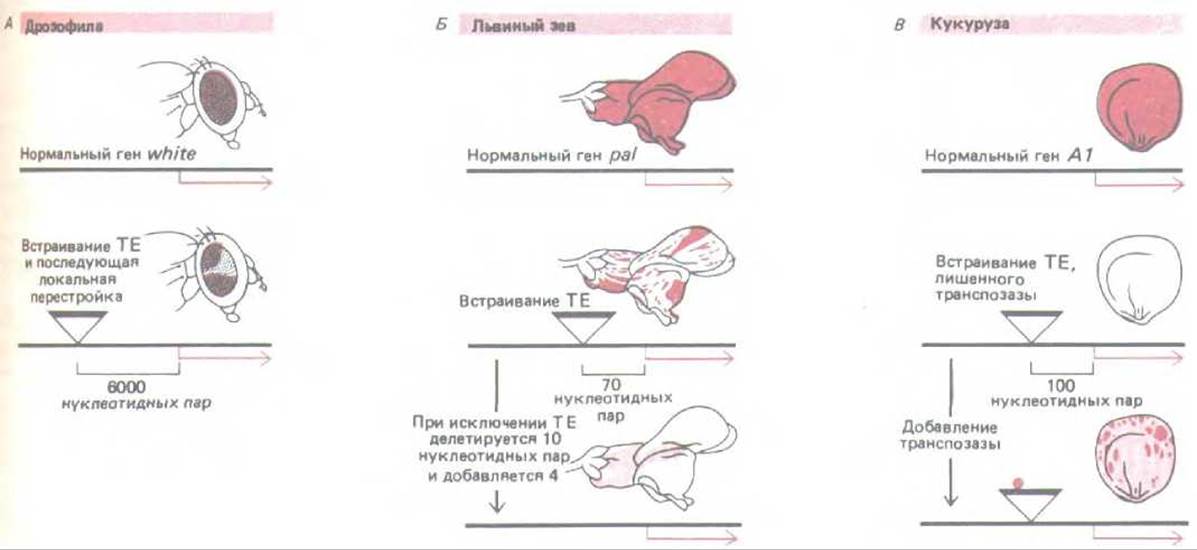
Рис. 10-72. Транспозоны могут обусловливать значительные изменения в регуляции генов. Для каждого из трех организмов приведен пример наследуемых изменений в распределении пигмента, вызванных встраиванием транспозонов (ТЕ) в регуляторные области генов. Аналогичные процессы могут приводить к морфологическим изменениям в организме, воздействуя на рост и дифференцировку клеток. А. Встраивание в регуляторные элементы, расположенные перед промотором гена white у дрозофилы, приводит к тому, что красный пигмент проявляется лишь в дорсальной и вентральной области глаза. Б. Встраивание перед промотором гена, определяющего образование пигмента у львиного зева, приводит к появлению цветов, у которых пегмент отсутствует везде, за исключением тех групп клеток, в которых этот элемент был удален при транспозиции. Последующее исключение транспозона из генома всего растения обусловливает появление на ограниченном участке цветка бледной окраски. В. Пример регулируемого изменения окраски зерна кукурузы, вызываемого транспозоном. В данном случае транспозон действует как регуляторный белок, отчасти восстанавливающий окраску во всех клетках зерна, которое в ином случае осталось бы неокрашенным. Кроме того, транспозаза катализирует случайное исключение элемента, в результате чего образуются отдельные интенсивно окрашенные пятна. (А -по G.M. Rubm et aL, Cold Spring Harbor Symp. Quant Biol. 50: 329-335 1985; Б-no E S Coen, R. Carpenter and C. Martin, Cell 47: 285-296,1986; B-no Zs. Schwarz-Sommer et at, EMBO J. 6: 287-294, 1987.)
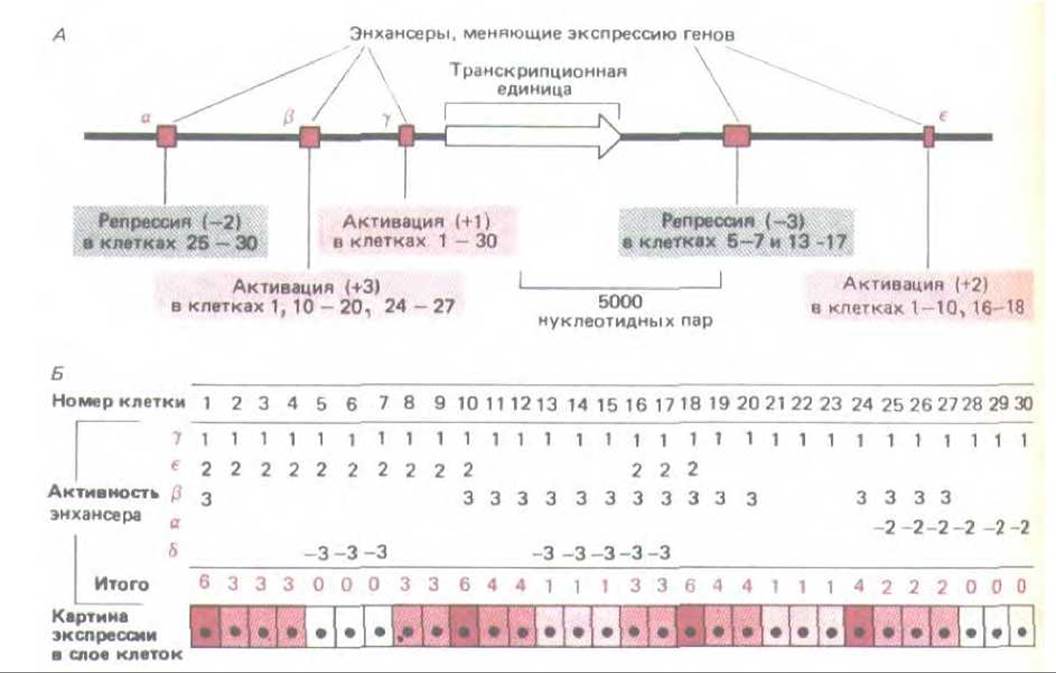
Рис. 10-73. Совместное действие отдельных энхансерных модулей приводит к характерному для каждой клетки порядку экспрессии генов. В связи с тем, что смесь регуляторных белков, связывающихся с каждым энхансером, варьирует от клетки к клетке, действие энхансера в различных клетках различно. Эта схема составлена по результатам, полученным на дрозофиле, где можно проанализировать много энхансеров, по-отдельности выявляя их действие в трансгенных мухах. Для простоты стимулирующее (+) и ингибируюшее (—) действие каждого энхансера (а, ß, y, δ, или ε) оценивают числами от +3 до -3 и считают, что эти числа можно складывать и вычислять суммарную активность энхансеров, определяющих уровень экспрессии генов.
Геномы высших эукариот, где длинные последовательности некодирующей ДНК перемежаются относительно короткими кодирующими участками, представляют собой «благодатную почву» для интеграции и исключения мобильных элементов. В связи с тем, что на транскрипцию генов влияют и удаленные от них на десятки тысяч нуклеотидных пар участки, можно ожидать, что многие возникшие при транспозиции изменения генома окажут влияние и на экспрессию генов. И напротив, по-видимому, лишь немногие перестройки приведут к разрушению коротких экзонов, содержащих кодирующие последовательности.
Может ли большой избыток некодирующей ДНК высших эукариот поддерживаться в ходе эволюции благодаря той регуляторной пластичности, которую она сообщает организму, содержащему много разнообразных транспозонов? То, что известно о регуляторных системах, контролирующих гены высших эукариот, находится в соответствии с этой возможностью. Энхансеры подобно экзонам, по-видимому, действуют как отдельные модули, и активность генов зависит от суммарного влияния на промотор набора энхансеров (рис. 10-73). Транспозоны, перемещая энхансеры по геному, могут способствовать оптимизации регуляции генов в целях выживания организма в ряду поколений.
10-36
10.5.10. Транспозиционные взрывы приводят к существенным изменениям в геномах и повышают биологическое разнообразие [62]
Другой уникальной особенностью, отличающей транспозоны от обычных мутагенов, является их способность долгое время находиться в состоянии покоя в хромосоме. Время от времени у части популяции бурно активируются движения транспозонов и, соответственно, их мутагенная активность. При таких катастрофических изменениях в геномах, называемых транспозиционными взрывами, происходит почти одновременное перемещение транспозонов нескольких типов. Впервые транспозиционные взрывы были обнаружены в развивающихся растениях кукурузы. Подобное явление наблюдается и при скрещивании определенных линий мух-феномен, известный под названием «гибридный дисгенез». Если такие взрывы происходят в клетках зародышевой линии, то они вызывают множественные изменения в геноме потомства отдельной мухи или растения.
Меняя свойства организма, транспозиционные взрывы повышают вероятность того, что два новых признака, каждый из которых сам по себе не обладает селективными преимуществами, окажутся выгодными, проявляясь вместе у единственной особи в популяции. Есть данные о том, что у определенных растений транспозиционные взрывы могут активироваться сильными стрессами, вызванными внешними условиями. Это приводит к появлению разнообразных, случайным образом модифицированных потомков, часть из которых может оказаться лучше приспособленной к выживанию в новых условиях. Возможно, что, по крайней мере у этих растений, существует механизм активации транспозонов, которые работают как мутагены и вызывают появление большого числа вариантных организмов в тот момент, когда такое разнообразие необходимо как никогда. Таким образом, транспозоны нельзя представлять себе только как паразитов; в некоторых случаях они способны действовать как полезные симбионты, способствуя выживанию тех видов, в геноме которых они содержатся.
10-37
10.5.11. Около 10% генома человека занимают два семейства транспозонов, которые, по-видимому, размножились лишь недавно [63]
ДНК приматов необычна по крайней мере в одном отношении: она содержит громадное количество копий двух последовательностей, про которые можно сказать, что ими прямо-таки «кишат» наши хромосомы. Оба типа этих последовательностей перемещаются в геноме в ходе РНК-опосредованного процесса, требующего обратную транскриптазу. Одна из этих последовательностей-L1-напоминает F-элемент у дрозофилы и сіn4-eлемент у кукурузы. Полагают, что она кодирует обратную транскриптазу (см. табл. 10-3). Транспозоны обычно возникают при участии систем контроля с обратной связью, которая жестко регулирует их число в каждой клетке (и таким образом спасает клетку от возможного бедствия); тем не менее, у человека L1-элементы составляют около 4% от всей массы генома.
Еще более необычная Alu-последовательность очень коротка (около 300 нуклеотидных пар) и перемещается, встраивая копию на месте сайта-мишени. Она образовалась в результате делеции гена 7SL-PHK хозяйской клетки. Этот ген кодирует РНК-компонент сигнал-узнающей частицы (SRP-signal-recognition particle), которая принимает участие в синтезе белка. Таким образом, неясно, следует ли рассматривать Alu-последовательность как транспозон или правильнее считать ее подвижным псевдогеном. Число копий Alu-последовательности в гаплоидном геноме человека составляет примерно 500000 (около 5% ДНК), таким образом, в среднем эта последовательность встречается один раз на каждые 5000 нуклеотидных пар ДНК. ДНК Alu транскрибируется с промотора 7SL-PHK, который узнается РНК-полимеразой III и находится внутри транскрипта. Следовательно, эта последовательность несет информацию, необходимую для своей собственной транскрипции.
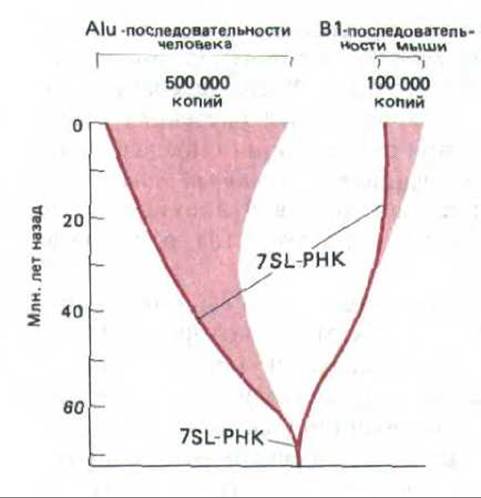
Рис. 10-74. Схема возможной эволюции повторяющихся Alu-подобных последовательностей, содержащихся в геномах мыши и человека. Полагают, что обе эти транспозирующиеся последовательности ДНК возникли из жизненно важного гена 7SL-РНК. Однако, принимая во внимание специфическое распределение и гомологию последовательностей этих высокоповторяющичся элементов, следует признать, что увеличение их копийности происходило независимо друг от друга.
Сравнение последовательности и расположения L1- и Alu-подобных элементов у различных млекопитающих позволяет сделать вывод, что эти элементы размножились и достигли высокой копийности относительно недавно (рис. 10-74). Трудно представить себе, что эти последовательности, рассеянные по всему геному, не оказывают заметного воздействия на близлежащие гены.
Заключение
Функциональные последовательности ДНК в геномах высших эукариот, по-видимому, собраны из небольших генетических модулей по крайней мере двух типов. Блоки кодирующих последовательностей образуют множество комбинаций для синтеза белков; регулирующие последовательности рассеяны среди длинных некодирующих участков и контролируют экспрессию генов. Как кодирующие последовательности (экзоны), так и регуляторные последовательности (энхансеры) по размеру обычно не превышают нескольких сот нуклеотидных пар. В геномах происходят разнообразные генетические рекомбинации, обусловливающие возникновение дупликацип и перенос последовательностей ДНК. В некоторых случаях дуплируются целые гены, которые могут затем приобретать новые функции. В результате рекомбинации иногда возникают новые белки, при этом происходит перетасовка экзонов или изменение экспрессии генов за счет перекомбинации энхансеров. Перестановка последовательностей имеет огромное значение для эволюции организмов, у эутриот она в значительной мере упрощена благодаря прерывистой структуре генов эукариот. Важно также, что гены эукариот подвержены многочисленным активирующим и подавляющим влияниям, которые оказывают на них разные комбинации удаленных от них энхансеров.
В геномах присутствуют различные типы транспозонов. Все вместе они составляют более 10% генома (и у дрозофилы, и у позвоночных). Время от времени в клетках зародышевой линии происходят «транспозиционные взрывы», приводящие ко многим наследуемым изменениям в экспрессии генов у одной и той же особи. Полагают, что транспозоны играют особую роль в эволюции, влияя на разнообразие организмов.
Литература
Общая
Lewin В. Genes, 3rd ed. New York, Wiley, 1987.
ScHteif R, Genetics and Molecular Biology. Reading M.A., Addison-Wesley, 1986.
Stem G. S. Molecular Genetics: An Introductory Narrative. San Francisco. Freeman, 1971.
Watson J.D., Hopkins N.H., Roberts J. W., Steitz J.A., Weiner A.M. Molecular Biology of the Gene, 4th ed. Menlo Park CA, Benjamin-Cummings, 1987.
Цитированная
1. Gurdon J. B. The developemental capacity of nuclei taken from intestinal epithelium cells of feeding tadpoles. J. Embryol. Exp. Morphol., 10, 622-640, 1962.
Steward F. C., Mapes M. O., Mears K. Growth and organized developement of cultured cells. Am. J. Bot, 45, 705-713, 1958.
2. Garrels J, I. Changes in protein synthesis during myogenesis in a clonal cell line. Dev. Biol., 73, 134-152, 1979.
3. Darned J. K, Jr. Variety in the level of gene control in eucaryotic cells. Nature, 297, 365-371, 1982.
Derman E. et al. Transcriptional control in the production of liver-specific mRNAs. Cell, 23, 731-739, 1981.
4. Gierer A. Molecular models and combinatorial principles in cell differentiation and morphogenesis. Cold Spring Harbor Symp. Quant. Bio)., 38, 951-961, 1974.
Scott M. P., OFanell P. H. Spatial programming of gene expression in early Drosophila embryogenesis. Annu. Rev. Cell Biol., 2, 49-80, 1986.
5. Maniatis Т., Goodbourn S., Fischer J.A. Regulation of inducible and tissue-specific gene expression. Science, 236, 1237-1244, 1987.
Yamamoto K. Steroid receptor regulated transcription of specific genes and gene networks. Annu. Rev. Genet; 19, 209-252, 1985.
6. Gehring W.J., Hiromi Y. Homeotic genes and homeobox. Annu. Rev. Genet; 20, 147-173, 1986.
7. Blau H.M. et al. Plasticity of the differentiated state. Science, 230, 758-766, 1985.
Davis R. L., Weintraub H., Lassar A. B. Expression of a single transfected cDNA converts fibrpblasts to myoblasts. Cell, 51, 987-1000, 1987.
8. Miller J. H., Reznikoff W.S., eds. The Operon. Cold Spring Harbor, NY. Cold Spring Harbor Laboratory, 1978.
Neidhardt F. C. et al., eds. Escherichia coli and Salmonella typhimurium: Cellular and Molecular Biology, Vol. 2, pp. 1439-1526. Washington DC. American Society for Microbiology, 1987. (Paradigms of operon regulation in bacteria.)
Ptashne M. A. Genetic Switch. Palo Alto, CA. Blackwell, 1986.
9. Gilbert W., Muller-Hill B. The lac operator in DNA. Proc. Natl. Acad, Sci. USA, 58, 2415-2421, 1967.
Gottesman S. Bacterial regulation: global regulatory networks. Annu. Rev. Genet., 18, 415-441, 1984.
Jacob F., Monod J. Genetic regulatory mechanisms in the synthesis of proteins. J. Mol. Biol; 3, 318-356, 1961.
Reznikoff W. S., Siegele D. A., Cowing D. W., Gross C. A. The regulation of transcription initiation in bacteria. Annu. Rev. Genet., 19, 355-388, 1985.
10. de Combrugghe В., Busby S., Вис Н. Cyclic AMP receptor protein: role in transcription activation. Science, 224, 831-838, 1984.
Hochschild A., Irwin M., Ptashne M. Represser structure and the mechanism of positive control. Cell, 32, 319-325, 1983.
Raibaud O., Schwartz M. Positive control of transcription initiation in bacteria. Annu. Rev. Genet., 18, 173-206, 1984.
11. Keener J., Wong P., Popham D., Wallis J., Kustu S. A sigma factor and auxiliary protiens required for nitrogen-regulated transcription in enteric bacteria. In: RNA Polymerase and the Regulation of Transcription. (W. S. Reznikoff et al, eds.), pp. 159-175. New York, Elsevier, 1987.
Ninfa A.J., Reitzer L.J., Magasanik B. Initiation of transcription at the bacterial у/иАр2 promoter by purified E. coli components is facilitated by enhancers. Cell, 50, 1039-1046, 1987.
12. Dunn T. M., Hahn S., Ogden S., Schlief R. F. An operator at -280 base pairs that is required for the repression of araBAD operon promoter. Proc. Natl. Acad. Sci. USA, 81, 5017-5020, 1984.
Griffith J., Hochschild A., Ptashne M. DNA loops induced by cooperative binding of lambda represser. Nature, 322, 750-752, 1986.
Mossing M. C., Record M. T. Upstream operators enhance repression of the lac promoter. Science, 233, 889-892, 1986.
13. Helmann J.D., Chamberlin M.J. Structure and function of bacterial sigma factors. Annu. Rev. Biochem; 57, 839-872, 1988.
14. Davidson B.L., Egly J.M., Mulvihill E. R., Chambon P. Formation of stable preinitiation complexes between eucaryotic class В transcription factors and promoter sequences. Nature, 301, 680-686, 1983.
Sawadogo M., Roeder R. G. Interaction of a gene-specific transcription factor with the adenovirus major late promoter upstream of the ТАТА box region. Cell, 43, 165-175, 1985.
Workman J. L., Roeder R. G. Binding of transcription factor TFIID to the major late promoter during in vitro nucleosome assembly potentiates subsequent initiation by RNA plymerase II. Cell, 51, 613-622, 1987. 15 Atchison M L. Enhancers: mechanisms of action and cell specificity. Annu. Rev. Cell Biol: 4, 127-153, 1988.
Maniatis Т., Goodbourn S., Fischer J. Regulation of inducible and tissue-specific gene expression. Science, 236, 1237-1245, 1987.
McKnight S.L., Kingsbury R. Transcriptional control signals of a eucaryotic protein-coding gene. Science, 217, 316-324, 1982.
Serfling E., Jasin M., Schaffner W. Enhancers and eukaryotic gene transcription. Trends Genet., 1, 224-230, 1985.
16. Emerson B. M., Nickol J. M., Jackson P. D., Felsenfeld G. Analysis of the tissue-specific enhancer at the 3' end of the chicken adult ß-globin gene. Proc. Natl. Acad. Sci. USA, 84, 4786-4790, 1987.
Evans Т., Reitman M., Felsenfeld G. An erythrocyte-specific DNA-binding factor recognizes a regulatory sequence common to all chicken globin genes. Proc. Natl. Acad. Sci. USA, 85, 5976-5980, 1988.
Jones N. C., Rigby P. W. J., Ziff E. B. Trews-acting protein factors and the regulation of eukaryotic transcription: lessons from studies on DNA tumor viruses. Cenes Dev., 2, 267-281, 1988.
Nomiyama H., Fromental C., Xiao J. H., Chambon P. Cell-specific activity of the constituent elements of Simian virus 40 enhancer. Proc. Natl. Acad. Sci. USA, 84 7881-7885, 1987.
17. Brent R., Ptashne M. A eukaryotic transcriptional activator bearing the DNA specificity of a prokaryotic represser. Cell, 43, 729-736, 1985. Evans R. M. The steroid and thyroid hormone receptor superfamily. Science, 240 889-895, 1988.
Kumar V. et al. Functional domains of the human estrogen receptor. Cell, 51 941-951, 1987.
Godowski P.J., Picard D., Yamamoto K. Signal transduction and transcriptionil regulation by glucocorticoid receptor - lexA fusion proteins. Science, 241, 812-816, 1988.
18. Sen R., Baltimore D. Inducibility of kappa immunoglobulin enhancer bindingprotein NF-kappa В by a posttranslational mechanism. Cell, 47, 921-928, 1986
Yamamoto K. K., Gonzalez G.A., Biggs W.H., MontminyM.R. Phosphorylationinduced binding and transcriptional efficacy of nuclear factor CREB. Nature, 334. 494-498, 1988.
Zimarino V., Wu C. Induction of sequence-specific binding of Drosophila heat shock activator protein without pritein synthesis. Nature, 327, 727-730, 1987.
19. Metzger D., White J. H., Chambon P. The human estrogen receptor functions in yeast. Nature, 334, 31-36, 1988.
Ptashne M. Gene regulation by proteins acting nearby and at a distance. Nature, 322, 697-701, 1986. Struhl K. Promoters, activator proteins, and the mechanism of transcriptional initiation in yeast. Cell, 49, 295-297, 1987.
20. Borst P., Greaves D. R. Programmed gene rearrangements altering gene expression. Science, 235, 658-667, 1987.
Mever T. F. Molecular basis of surface antigen variation in Neisseria. Trends Genet, 3, 319-324, 1987.
Simon M., Zieg J., Silverman M., Mandel G., Doolittle R. Phase variation: evolution of a controlling element. Science, 209, 1370-1374, 1980.
21. Cross F., Hartwell L.H., Jackson C., Konopka J.B. Conjugation in Saccharomyas cerevisiae. Annu. Rev. Cell Biol; 4, 429-457, 1988. Herskowitz I. Master regulatory loci in yeast and lambda. Cold Spring Harbor Symp. Quant. Biol., 50, 565-574, 1985.
Kushner P. J., Blair L. C., Herskowitz I. Control of yeast cell types by mobile genes: a test. Proc. Natl. Acad. Sci. USA, 76, 5264-5268, 1979.
22. Kostriken R., Strathern J. N.. Klar A., Hicks J. В., Hefforn F. A site-specific endonuclease essential for mating-type switching in Saccharomyces cerevisiae. Cell, 35, 167-174, 1983.
23. Nasmyth K., Shore D. Transcriptional regulation in the yeast life cycle. Science, 237, 1162-1170, 1987.
24. Brand A. H., Breeden L., Abraham J., Sternglanz R., Nasmyth K. Characterization of the "silencer" in yeast: a DNA sequence with properties opposite to those of a transcriptional enhancer. Cell, 41, 41-48, 1985.
25. Friedman D. I. et al. Interactions of bacteriophage and host macromolecules in the growth of bacteriophage lambda. Microbiol. Rev., 48, 299-325, 1984.
Ptashne M. et al. How the lambda represser and его work. Cell, 19, 1-11, 1980.
26. Brown D. D. The role of stable complexes that repress and activate eukaryotic genes. Cell, 37, 359-365, 1984.
Weintraub H. Assembly and propagation of repressed and derepressed chromosomal states. Cell, 42, 705-711, 1985.
27. Brown S.W. Heterochromatin. Science, 151, 417-425, 1966.
Hsu T. C., Cooper J. E. K., Mace M. L., Brinkley B. R. Arrangement of centromeres in mouse cells. Chromosoma, 34, 73-87, 1971.
28. Gartler S. M., Riggs A. D. Mammalian X-chromosome inactivation. Annu. Rev. Genet, 17, 155-190, 1983.
Lock L. F., Takagi N., Martin G. R. Methylation of the Hprt gene on the inactive X occurs after chromosome inactivation. Cell, 48, 39-46, 1987.
LyonM. F. X-chromosome inactivation and developmental patterns in mammals. Biol. Rev., 47, 1-35, 1972.
29. Baker W.K. Position-effect variegation. Adv. Genet, 14, 133-169, 1968.
Spofford J. B. Position-effect variegation in Drosophila. In: The Genetics and Biology of Drosophila (M. Ashburner, E. Novitski, eds.), Vol. 1C, pp. 955-1018. New York, Academic Press, 1976.
30. Goldberg D. A., Posakony J. W., Maniatis T. Correct developmental expression of a cloned alcohol dehydrogenase gene transduced into the Drosophila germ line. Cell, 34, 59-73, 1983. Grosveld F., van Assendelft G. В., Greaves D. R., Kollias G. Position independent, high-level expression of the human ß-globin gene in transgenic mice. Cell, 51, 975-985, 1987.
Meyerowitz E. M., Raghavan K. V., Mothers P. H., Roark M. How Drosophila larvae make glue: control of Sc/S-3 gene expression. Trends Genet., 3, 288-293, 1987.
Palmiter R. D., Brinster R. L. Germ-line transformation of mice. Annu. Rev. Genet., 20, 465-499, 1986.
31. Ephrussi A., Church G.M., Tonegawa S., Gilbert W. В lineage-specific interactions of an immunoglobulin enhancer with cellular factors in vivo. Science, 227, 134-140, 1985.
Garel A., Zolan M., Axel R. Genes transcribed at diverse rates have a similar conformation in chromatin. Proc. Mail. Acad. Sci. USA, 74, 48674871, 1977.
Karlsson S., Nienhuis A. W. Developmental regulation of human globin genes. Annu. Rev. Biochem., 54, 1071-1108, 1985.
Weintraub H., Groudine M. Chromosomal subunits in active genes have an altered conformation. Science, 193, 848-856, 1976.
32. Brill S.J., Sternglanz R. Transcription-dependent DNA supercoiling in yeast DNA topoisomerase mutants. Cell, 54, 403-411, 1988.
Wang J.C. Superhelical DNA. Trends Biochem. Sci; 5, 219-221, 1980. Wanq ]. C., Giaever G. N. Action at a distance along a DNA. Science, 240, 300-304, 1988.
33. GuarenteL. Regulatory proteins in yeast. Annu. Rev. Genet., 21, 425-452, 1987.
34. Razin A., Cedar H., Riggs A.D., eds. DNA Methylation: Biochemistry and Biological Significance. New York, Springer-Verlag, 1984.
35. Cedar H. DNA methylation and gene activity. Cell, 53, 3-4, 1988.
Ivarie R, D., Schacter B. S., O'Farrell P. H. The level of expression of the rat growth hormone gene in liver tumor cells is at least eight orders of magnitude less than that in anterior pituitary cells. Мої. Cell. Biol., 3, 1460-1467, 1983.
Yisraeli J. et al. Muscle-specific activation of a methylated chimeric actin gene. Cell, 46, 409-416, 1986.
36. Bird A. P. CpG islands as gene markers in the vertebrate nucleus. Trends Genet, 3, 342-347, 1987.
37. Duncan L The bithorax complex. Annu. Rev. Genet; 21, 285-319, 1987.
Peifer M., Karchi F., Bender W. The bithorax complex: control of segmental identity. Genes Dev; 1, 891-898, 1987
38. Landrick R., Yanofsky C. Transcription attenuation. In; Escherichia coli and Salmonella typhimurium: Cellular and Molecular Biology (F.C. Neihardt et al, eds.), Vol. 2, pp. 1276 1301, Washington DC, American Society for Microbiology, 1987.
Plan T. Transcription termination and the regulation of gene expression. Annu. Rev. Biochem., 55, 339-372, 1986.
Yanofsky C. Operon-specific control by transcription attenuation. Trends Genet., 3, 356-360, 1987.
39. Andreadis A., Gallego M.E., Nadal-Ginard B. Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications. Annu. Rev. Cell Biol; 3, 207-242, 1987.
Leff S., Rosenfeld M., Evans R. Complex transcriptional units: diversity in gene expression by alternative RNA processing. Annu. Rev. Biochem; 55, 1091-1117, 1986.
Schwarz T. L., Tempel B. L., Papazian D. M., Jan Y. N., Jan L. Y. Multiple potassium-channel components are produced by alternative splicing at the Shaker locus in Drosophila. Nature, 331, 137-142, 1988.
40. Baker B. S., Belote J. M. Sex determination and dosage compensation in Drosophila melanogaster. Annu. Rev. Genet., 17, 345-393, 1983. Bingham P. M., Chou Т., Mims L, Zachar Z. On/off regulation of gene expression at the level of splicing. Trends Genet. 4, 134-138, 1988. Boggs R. Т., Gregor P., Idriss S., Belote J. M., McKeown M. Regulation of sexual differentiation in Drosophila melanogaster via alternative splicing of RNA from the transformer gene. Cell, 50, 739-747, 1987.
Laski F. A., Rio D. C., RubinG.M. Tissue specificity of Drosophila P element transposition is regulated at the level of RNA splicing. Cell, 44, 7-19, 1986.
41. Early P. et al. Two mRNAs can be produced from a single immunoglobulin p gene by alternative RNA processing pathways. Cell, 20, 313-319, 1980.
Peterson M. L., Perry R. P. Regulated production of μm and μs mRNA requires linkage of the poly(A) addition sites and is dependent on the length of the μm - μs intron. Proc. Natl. Acad. Sci. USA, 83, 8883-8887, 1986.
42. Beadle G. Genes and the chemistry of the organism. Am. Sci., 34, 31-53, 1946.
43. Newport J.W., Forbes D.J. The nucleus: structure, function, and dynamics. Annu. Rev. Biochem., 56, 535-565, 1987.
Schneider R.J., Shenk T. Impact of virus infection on host cell protein synthesis. Annu. Rev. Biochem; 56, 317-332, 1987.
44. Aziz N.; Munro H. N. Intron regulates ferritin mRNA translation through a segment of its 5' untranslated region. Proc. Natl. Acad. Sci. USA, 84, 8478-8482, 1987.
Gold L. Posttanscriptional regulatory mechanisms in Escherichia coli. Annu. Rev. Biochem., 57, 199-234, 1988.
Nomura M., Course R., Baughman G. Regulation of the synthesis of ribosomes and ribosomal components. Annu. Rev. Biochem; 53, 75-117, 1984.
Walden W. E. et al. Translational repression in eucaryotes: partial purification and characterization of a represser of ferritin in RNA translation. Proc. Natl. Acad. Sci. USA, 85, 9503-9507, 1988.
45. Hunt T. False starts in translational control of gene expression. Nature, 316, 580-581, 1985.
Kozak M. Bifunctional messenger RNAs in eucaryotes. Cell, 47, 481-483, 1986.
Pelletier J., Sonenberg N. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature, 334, 320-325, 1988.
46. Ilan J., ed. Translational Regulation of Gene Expression. New York, Plenum Press, 1987.
Rosenthal E. Т., Hunt Т., Ruderman J. V. Selective translation of mRNA controls the pattern of protein synthesis during early development of the surf clam, Spisula solidissima. Cell, 20, 487-494, 1980.
Walden W.E., Thach R.E. Translalional control of gene expression in a normal fibroblast: characterization of a subclass of mRNAs with unusual kinetic properties. Biochemistry, 25, 2033-2041, 1986.
47. Craigen W. J., Caskey C. T. Translational frameshifting: where will it stop? Cell, 50, 1-2, 1987.
48. Casey J. L. et al. Iron-responsive elements: regulatory RNA sequences that control mRNA levels and translation. Science, 240, 924-928, 1988. Raghow R. Regulation of messenger RNA turnover in eukaryotes. Trends Biochem. Sci., 12, 358-360, 1987.
Shaw G., Kamen R.A. conserved AU sequence from the 3' untranslated region of GM-CSF mRNA mediaters selective mRNA degradation. Cell, 46, 659-667, 1986.
49. Graves R.A., Pandey N.B., Chodchoy N., Marzluff W.F. Translation is required for regulation of histone mRNA degradation. Cell, 48, 615-626, 1987.
Marzluff W.F., Pandey N. B. Multiple regulatory steps control histone mRNA concentrations. Trends Biochem. Sci., 13, 49-52, 1988.
Mowry K. L., Steits J. A. Identification of the human U7 snRNP as one of several factors involved in the 3' end maturation of histone premessenger RNAs. Science, 238, 1682-1687, 1987.
50. Driever W., Nusslein-Volhard C.A. gradient of bicoid protein in Drosophila embryos. Cell, 54, 83-93, 1988.
Lawrence J. В., Singer R. H. Intracellular localization of messenger RNAs for cytoskeletal protiens. Cell, 45, 407-415, 1986.
Weeks D.L., Me/ton D.A. A maternal mRNA localized to the vegetal hemisphere in Xenopus eggs codes for a growth factor related to TGF- beta. Cell, 51, 861-867, 1987.
51. Borst P. Discontinous transcription and antigenic variation in trypanosomes. Annu. Rev. Biochem., 55, 701-732, 1986.
Eisen H. RNA editing: who's on first? Cell, 53, 331-332, 1988.
Powell L. M. et al. A novel form of tissue-specific RNA processing produces apolipoprotein-B48 in intestine. Cell, 50, 831-840, 1987. Sharp P. A. Trans splicing: variation on a familiar theme. Cell, 50, 147-148, 1987.
52. McClain W. H., Guerrier-Takada C., Altman S. Model substrates for an RNA enzyme. Science, 238, 527-530, 1987.
Pine O., Inouye M. Antisense RNA regulation in prokaryotes. Trends Genet; 2, 284-287, 1986.
Tomizawa J. Control of ColEI plasmid replication: binding of RNA I to RNA II and inhibition of primer formation. Cell, 47, 89-97, 1986. Wu H. N., Uhlenbeck О. С. Role of a bulged A residue in a specific RNA-protein interaction. Biochemistry, 26, 8221-8227, 1987.
53. Clarke B.C., Robertson A., Jeffreys A.J., eds. The Evolution of DNA Sequences. London. The Royal Society, 1986.
Nei M., Koehn R. K., eds. Evolution of Genes and Proteins. Sunderland MA, Sinauer, 1983.
54. Doolittle R.F., Proteins. Sci. Am; 253 (4), 88-99, 1985.
Holland S. K., Blake C. C. Proteins, exons, and molecular evolution. Biosystems, 20, 181-206, 1987.
Maeda N.. Smithies O. The evolution of multigene families: human haptoglobin gene. Annu. Rev. Genet., 20, 81-108, 1986.
55. Kourilsky P. Molecular mechanisms for gene conversion in higher cells. Trends Genet., 2, 60-63, 1986.
Roth D. В., Porter Т. N.. "Wilson J. Н. Mechanisms of uonbomologous recombination in cells. Мої. Cell. Biol; 5, 2599-2607, 1985.
Smith G. P. Evolution of repeated DNA sequences by unequal crossovers. Science, 191, 528-535, 1976.
Stark G.R., Wahl G.M. Gene amplification. Annu. Rev. Biochem., 53, 447-491, 1984.
56. Dickerson R. E., Geis I. Hemoglobin: Structure, Function, Evolution and Pathology. Menlo Park CA, Benjamin-Cummings, 1983.
Efstratiadis A. et al. The structure and evolution of the human p-globin gene family. Cell, 21, 653-668, 1980.
Vollrath D., Nathans J., Davis R. W. Tandem array of human visual pigment genes at Xq28. Science, 240, 1669-1672, 1988.
57. Doolittle W.F. RNA mediated gene conversion? Trends Genet., 1, 64-65, 1985.
Gilbert W., Marchionni M., McKnight G. On the antiquity of introns. Cell, 46, 151-153, 1986.
Sharp P. On the origin of RNA splicing and introns. Cell, 42, 397-400, 1985.
58. Britten R.J., Kohne D.E. Repeated sequences in DNA. Science, 161, 529-540, 1968.
Jelineh W. R., Schmid C. W. Repetitive sequences in eukaryotic DNA and their expression. Annu. Rev. Biochem; 51, 813-844, 1982.
59. Craig-Holmes A. P., Shaw M. W. Polymorphism of human constitutive heterochromatin. Science, 174, 702-704, 1971.
Hsu Т. С. Human and Mammalian Cytogenetics: A Historical Perspective. New York, Springer-Verlag, 1979.
John В., Miklos G.L.G. Functional aspects of satellite DNA and heterochromatin. Int. Rev. Cytol; 58, 1-114, 1979.
Orgel L. E., Crick F. H. C. Selfish DNA: the ultimate parasite. Nature, 284, 604-607, 1980.
60. Berg D. E., Howe M. M., eds. Mobile DNA. Washington DC, American Society for Microbiology, 1989. Daring H.-P., Starlinger P. Molecular genetics of transposable elements in plants. Annu. Rev. Genet., 20, 175-200, 1986.
Finnegan D.J. Transposable elements in eukaryotes. Int. Rev. Cytol., 93, 281-326, 1985.
McCIintock B. Controlling elements and the gene. Cold Spring Harbor Symp. Quant. Biol., 21, 197-216, 1956.
61. Coen E.S., Carpenter, R. Transposable elements in Antirrhinum majus: generators of genetic diversity. Trends Genet; 2, 292-296, 1986. Georgiev G. P. Mobile genetic elements in animal cells and their biological significance. Eur. J. Biochem., 145, 203-220, 1984.
O'Kane C.J., Gehring W. Detection in situ of genomic regulatory elements in Drosophila, Proc. Natl. Acad. Sci. USA, 84, 9123-9127, 1987. Hiromi Y., Gehring W.J. Regulation and function of the Drosophila segmentation gene/MS/I/ tarazu. Cell, 50, 963-974, 1987.
62. Gerasimova Т.1., Mizrokhi L.J., Georgiev G.P. Transposition bursts in genetically unstable Drosophila. Nature, 309, 714-716, 1984. McCIintock B. The significance of responses of the genome to challenge. Science, 226, 792-801, 1984.
Walbot V., Cullis C.A. Rapid genomic change in higher plants. Annu. Rev. Plant Physiol., 36, 367-396, 1985.
63. Deininger P. L., Daniels G. R. The recent evolution of mammalian repetitive DNA elements. Trands Genet; 2, 76-80, 1986.
Ruffner D. E., Sprung C. N.. Minghetti P. P., Gibbs P. E., Dugiaczyk A. Invasion of the human albumin-a-fetoprotein gene family by Alu, Kpn, and two novel repetitive DNA elements. Мої. Biol. Evol; 4, 1-9, 1987.
Weiner A. M., Deininger P. L., Efstratiadis A. Nonviral retroposons: genes, pseudo-genes, and transposable elements generated by the reverse flow of genetic information. Annu. Rev. Biochem; 55, 631-661, 1986.