Молекулярная биология клетки - Том 1 - Албертс Б., Брей Д., Льюис Дж., Рэфф М., Робертс К., Уотсон Дж. 1994
Молекулярная организация клеток
Основные генетические механизмы
Механизмы репликации ДНК
Живые организмы должны не только поддерживать целостность нуклеотидных последовательностей ДНК путем ее репарации, но еще и очень точно воспроизводить свою ДНК перед каждым клеточным делением. При репликации ДНК скорость полимеризации колеблется в пределах от 500 нуклеотидов в 1 с у бактерий приблизительно до 50 нуклеотидов у млекопитающих. Ясно, что ферменты, катализирующие процесс репликации, должны работать и точно, и быстро. Быстрота и точность достигаются с помощью особого мультиферментного комплекса, направляющего процесс репликации. Этот комплекс, состоящий из нескольких различных белков, представляет собой сложный и совершенный «аппарат репликации».
5.3.1. Репликация ДНК, как и ее репарация, основана на комплементарном спаривании оснований [25]
Матричная активность ДНК проявляется в том, что ее нуклеотидная последовательность копируется (целиком или частично) путем комплементарного спаривания оснований (А с Т или G с С) в виде комплементарной последовательности нуклеотидов ДНК или РНК. Этот процесс предполагает узнавание каждого нуклеотида в ДНК свободным (неполимеризованным) комплементарным нуклеотидом и обязательное разделение (хотя бы на время) двух цепей ДНК, с тем чтобы в каждом основании группы, играющие роль доноров и акцепторов при образовании водородных связей, оказались доступными для комплементарного спаривания. Таким образом поступающие одиночные нуклеотиды выстраиваются в определенном порядке вдоль матричной цепи ДНК для ферментативной полимеризации, продуктом которой является новая полинуклеотидная цепь. В 1957 г. был открыт первый фермент, катализирующий процесс полимеризации нуклеотидов; он был назван ДНК-полимеразой. Было показано, что субстратами ДНК-полимеразы служат дезоксирибонуклеозидтрифосфаты, полимеризующиеся на одно-цепочечной ДНК-матрице (двухступенчатый механизм этой полимеризации представлен на рис. 5-34 в связи с обсуждением процесса репарации ДНК). Позже была выделена и РНК-полимераза, для которой субстратами служат рибонуклеозидтрифосфаты.
Во время репликации ДНК каждая из двух ее старых цепей служит матрицей для образования новой цепи. Поэтому чрезвычайно длинная нуклеотидная последовательность клеточной ДНК реплицируется, как это принято называть, «полуконсервативно» и каждая из двух дочерних клеток получает при клеточном делении новую двойную спираль ДНК, состоящую из одной старой и одной новой цепи (см. рис. 3-11).
5-23
5.3.2. Репликационная вилка асимметрична [26]
Исследования, проведенные в начале 1960-х годов на реплицирующихся хромосомах, в которые в качестве импульсной метки вводили радиоактивный предшественник ДНК 3Н-тимидин, выявили особую четко ограниченную область репликации, перемещающуюся вдоль родительской спирали ДНК. Эта активная область из-за своей Y-образной формы была названа репликационной вилкой. Именно в ней с помощью мультиферментного комплекса, содержащего ДНК-полимеразу, синтезируются дочерние молекулы ДНК.
В то время казалось вполне вероятным, что простейший механизм репликации ДНК заключается в непрерывном росте обеих новых цепей нуклеотид за нуклеотидом по мере перемещения репликационной вилки от одного конца молекулы ДНК к другому. Однако, поскольку две цепи в спирали ДНК антипараллельны, одна из дочерних цепей должна расти в направлении 5' → 3', а другая - в направлении 3' → 5'. В таком случае репликационной вилке потребовалось бы две разные ДНК-полимеразы. Одна из них наращивала бы цепь в направлении 5' → 3' (рис. 5-34); при этом каждый поступающий мономер (дезоксирибонуклеозидтрифосфат) приносит с собой необходимую для его присоединения к цепи энергию (ее носителем является трифосфатная группа). Другая ДНК-полимераза, перемещающаяся в направлении 3' → 5', должна катализировать «рост с головы»; в этом случае энергию, необходимую для присоединения каждого очередного нуклеотида, должен нести конец растущей цепи ДНК. В действительности такой (3' → 5') ДНК-полимеразы не существует (рис. 5-38), хотя биохимикам известны некоторые другие процессы полимеризации, протекающие по типу «роста с головы» (см. рис. 2-34), Каким же образом происходит рост цепи в направлении 3' → 5'? Возможный ответ на этот вопрос подсказали в конце 1960-х годов эксперименты с радиоактивно меченными предшественниками ДНК. Если растущие клетки получают всего на несколько секунд высокорадиоактивный 3Н-тимидин, то метка включается лишь в ДНК, синтезированную в самый последний момент, т. е. в ту ее часть, которая следует непосредственно за репликационной вилкой. Этим методом избирательного введения метки было выявлено, что при репликации бактериальной ДНК в области репликационной вилки образуются и какое-то время существуют фрагменты, насчитывающие от 1000 до 2000 нуклеотидов (впоследствии за ними закрепилось название «фрагменты Оказаки»; у эукариот они гораздо короче: от 100 до 200 нуклеотидов). Несколько позже было показано, что синтез этих фрагментов ДНК идет только в направлении 5' → 3'; синтезированные фрагменты соединяются затем в длинные цепи ДНК под действием того же фермента, который сшивает разрывы в спирали ДНК во время ее репарации, т.е. под действием ДНК-лигазы (см. рис. 5-35).
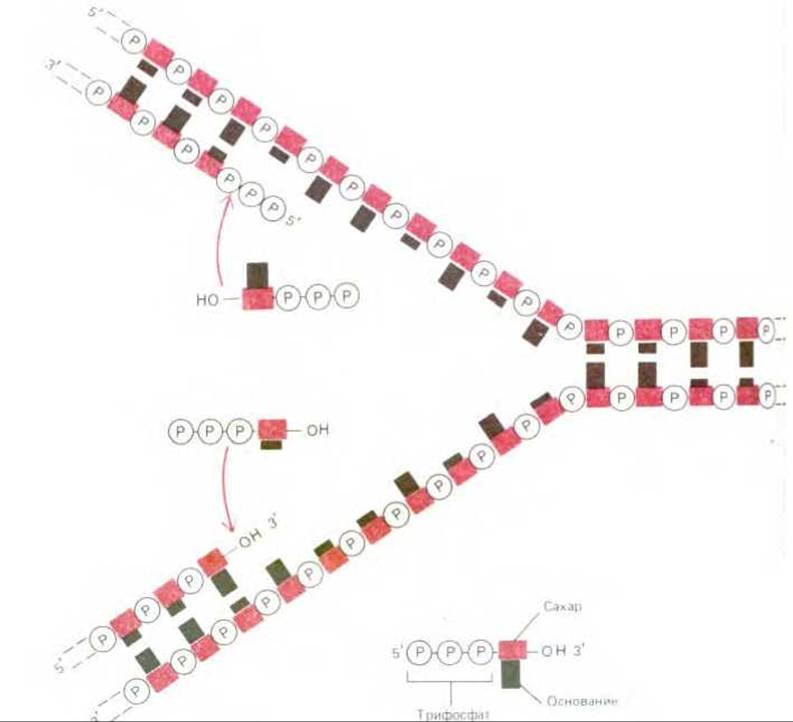
Рис. 5-38. На первый взгляд простейшим механизмом репликации ДНК представляется механизм, изображенный на этой (неверной!) схеме. Обе дочерние цепи должны были бы при этом расти непрерывно за счет присоединения нуклеотидов соответственно в 5' → 3' - направлении (на рисунке - внизу) и 3' → 5' - направлении (на рисунке - вверху). Однако фермента, который бы катализировал присоединение нуклеотидов в направлении 3' → 5', не существует.
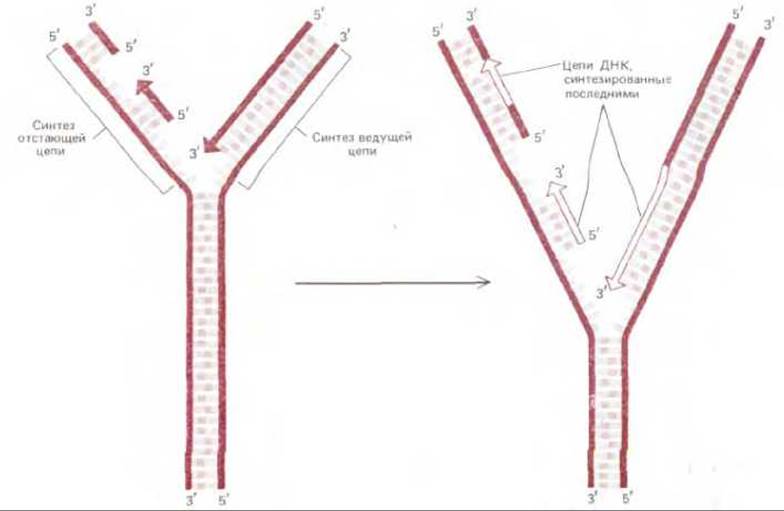
Рис. 5-39. Строение репликационной вилки. Обе дочерние цепи строятся в направлении 5' → 3'. Для этого отстающая цепь ДНК должна синтезироваться в виде ряда коротких фрагментов (фрагменты Оказаки).
Репликационная вилка асимметрична (рис. 5-39). Из двух синтезируемых дочерних цепей ДНК одна строится непрерывной, а другая прерывистой. Первую называют ведущей (или лидирующей), а вторую - отстающей. Наращивание второй цепи отстает, потому что образование каждого фрагмента Оказаки оказывается возможным лишь после того, как продвижение ведущей цепи откроет соответствующий участок матрицы. Хотя в целом вся эта цепь строится в направлении 3' → 5', каждый из ее фрагментов синтезируется в направлении 5' → 3'. Благодаря тому что ДНК на отстающей части вилки строится при помощи механизма, работающего прерывисто по типу «шитья назад иголкой» (backstitching), в репликационной вилке не требуется никакого другого фермента, кроме (5' → 3')-ДНК-полимеразы.
5-24
5.3.3. Высокая точность репликации ДНК предполагает наличие механизма, осуществляющего коррекцию [27]
Точность копирования при репликации ДНК столь велика, что в среднем на каждые 1-109 комплементарных пар, образующихся в процессе воспроизведения генома млекопитающих, насчитывающего 3-104 пар оснований (см. разд. 9.1.3), приходится приблизительно одна ошибка. Точность эта значительно превосходит ту, какую следует ожидать, учитывая, что во время репликации образуются не только обычные комплементарные пары оснований. В нормальной ДНК возникают на короткое время с частотой 10-4-10-5 редкие таутомерные формы всех четырех ее оснований. Эти формы образуют неправильные пары. Так, редкая таутомерная форма С спаривается с А вместо G, в результате чего возникает мутация (рис. 5-40). Таким образом высокая точность репликации ДНК определяется наличием механизмов, осуществляющих коррекцию, т. е. устраняющих подобные ошибки.
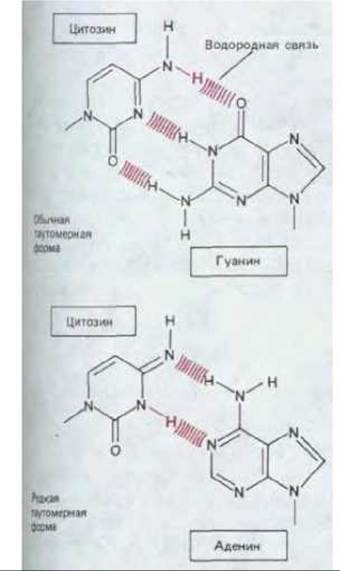
Рис. 5-40. Пример возникновения при репликации ДНК неправильной пары оснований: находясь в термодинамически невыгодной таутомерной форме, цитозин легко образует водородные связи с аденином.

Рис. 5-41. Схема, поясняющая, как протекает процесс коррекции (устранение ошибок) при синтезе ДНК, катализируемом ДНК-полимеразами у бактерий. Предполагается, что аналогичный механизм коррекции действует и в эукариотических клетках.
Один из важных механизмов коррекции зависит от особых свойств ДНК-полимеразы. В отличие от РНК-полимераз ДНК-полимеразы не могут начать синтез новой полинуклеотидной цепи, просто связав друг с другом 3'-ОН- конец какой-либо полинуклеотидной цепи, которая должна быть спарена с матричной цепью ДНК; ДНК-полимеразы способны только добавлять новые нуклеотиды к уже имеющемуся 3'-ОН-концу полинуклеотидной цепи (см. рис. 534). Эту предобразованную цепь, к которой добавляются нуклеотиды, называют затравкой или праймером. Молекулы ДНК с затравкой, у которой 3'-ОН-конец не спарен, не могут служить матрицами. Бактериальные ДНК-полимеразы способны, однако, с ними работать. Вступив в контакт с такими молекулами ДНК, они используют присущую им (3' → 5')-экзонуклеазную активность и отщепляют (путем гидролиза) любые неспаренные нуклеотиды на затравочном конце. Отщепляется ровно столько нуклеотидов, сколько требуется для того, чтобы у затравки появился спаренный конец и образовалась активная матрица. Действуя таким образом, ДНК-полимераза выступает в роли «самокорректирующего» фермента: она устраняет свои собственные ошибки, возникающие в процессе полимеризации. Рис. 5-41 поясняет, как этот тип коррекции может использоваться для удаления неправильных пар С—А, образуемых редкой таутомерной формой цитозина.
Потребность в правильно спаренном конце как раз и наделяет ДНК- полимеразу способностью исправлять свои собственные ошибки. Такой фермент, очевидно, мог бы начать синтез ДНК при полном отсутствии затравки, только утратив способность различать спаренный и неспаренный концы. В то же время РНК-полимеразы, участвующие в транскрипции генов (см. разд. 5.1.1), судя по всему не нуждаются в самокоррекции, потому что ошибки транскрипции не передаются следующему поколению и случайно возникшие дефектные молекулы особой роли не играют. РНК-полимеразы могут начинать синтез новых полинуклеотидных цепей в отсутствие затравки, причем ошибки встречаются с частотой 10-4 как при синтезе РНК, так и при трансляции, т.е. при переводе нуклеотидных последовательностей мРНК в аминокислотные последовательности белков.
5-22
5.3.4. Репликация ДНК в направлении 5’ → 3’ обеспечивает эффективную коррекцию
Весьма вероятно, что однонаправленность репликации ДНК (5' → 3') объясняется высокими требованиями к точности процесса. Если бы существовала ДНК-полимераза, присоединяющая дезоксирибонуклеозидтрифосфаты к синтезируемой полинуклеотидной цепи таким образом, что эта цепь росла в направлении 3' → 5', то активирующую трифосфатную группировку нес бы растущий 5'-конец цепи, а не поступающий мононуклеотид. В этом случае ошибки полимеризации не могли бы устраняться простым гидролизом, потому что появление свободного 5'-конца немедленно обрывало бы синтез ДНК. Ясно, что основание, только что неправильно спарившееся на 3'-конце, устранить гораздо легче, чем такое же основание, присоединившееся к 5'-концу цепи ДНК. Поэтому, хотя механизм репликации ДНК, изображенный на рис. 5-39, кажется на первый взгляд значительно более сложным и громоздким, чем неверный гипотетический механизм, представленный на рис. 5-38, этот реально функционирующий механизм способен обеспечить гораздо большую точность именно в силу того, что синтез ДНК идет здесь только в направлении 5' → 3'.
5-25
5.3.5. Для синтеза коротких затравочных молекул на матрице отстающей цепи требуется особый фермент [28]
С того момента, как возникла репликационная вилка, для ДНК-полимеразы, синтезирующей ведущую цепь, всегда есть спаренный 3'-конец, необходимый ей для того, чтобы начать синтез новой цепи. Иначе обстоит дело с ДНК-полимеразой, ответственной за синтез отстающей цепи. Ей требуется всего каких-нибудь 4 с для того, чтобы синтезировать один короткий фрагмент ДНК, после чего она должна переключиться на синтез совсем другого фермента на новом участке матричной цепи, расположенной на некотором расстоянии от первого (см. рис. 5-39). Для этого ей всякий раз нужна затравка со спаренным 3'-концом, а следовательно, нужен и механизм, способный производить такие затравки. В этот механизм входит фермент, называемый ДНК-праймазой. Она синтезирует из рибонуклеозидтрифосфатов короткие РНК-затравки (праймеры), состоящие у эукариот примерно из 10 нуклеотидов (рис. 5-42). Эти затравки синтезируются с определенными интервалами на матрице для отстающей цепи; здесь их наращивает ДНК-полимераза, начиная, таким образом, всякий раз новый фрагмент Оказаки. Молекула ДНК-полимеразы продолжает это наращивание до тех пор, пока она не достигнет РНК-затравки, присоединенной к 5'-концу предыдущего фрагмента ДНК. Чтобы обеспечить образование непрерывной цепи ДНК из многих таких фрагментов, в действие вступает особая система репарации ДНК, быстро удаляющая РНК-затравку и заменяющая ее на ДНК. Завершает процесс ДНК-лигаза, соединяющая 3'-конец нового фрагмента ДНК с 5'-концом предыдущего фрагмента (рис. 5-43).
Почему предпочтение отдается удаляемой РНК-затравке, а не ДНК-затравке, которую не требовалось бы удалять? Выше мы отмечали, что самокорректирующая полимераза не способна начинать синтез полинуклеотидных цепей de novo; это предполагает и обратное утверждение: тот фермент, который начинает синтез цепей de novo, к эффективной самокоррекции не способен. Значит, любой фермент, катализирующий инициацию синтеза фрагментов Оказаки, неизбежно создал бы не слишком точную копию (не менее 1 ошибки на 105). Это означало бы колоссальное увеличение частоты мутаций даже при том, что количество таких копий, сохранившееся в конечном продукте, составляло бы не более 5% всего генома (например, 10 нуклеотидов во фрагменте, состоящем из 200 нуклеотидов). Естественно думать поэтому, что выдвижение РНК, а не ДНК на роль затравки обеспечивало важное преимущество, поскольку рибонуклеотиды автоматически метят такие последовательности, как «плохие копии», которые должны быть удалены.
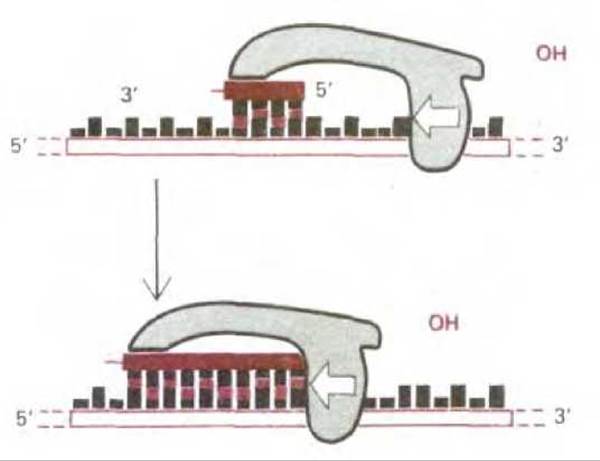
Рис. 5-42. Схема реакции, катализируемой праймазой - ферментом, синтезирующим короткие РНК-затравки в отстающей цепи ДНК. В отличие от ДНК-полимеразы этот фермент способен начинать синтез новой полинуклеотидной цепи с соединения двух нуклеозидтрифосфатов. Образовав короткий полинуклеотид, праймаза прекращает работу. Теперь к свободному 3'-концу может добавлять нуклеотиды ДНК-полимераза.
5-26
5-33
5.3.6. Особые белки способствуют расплетанию двойной спирали ДНК перед репликационной вилкой [29]
Двойная спираль ДНК должна расплетаться по ходу продвижения репликационной вилки, для того чтобы поступающие дезоксирибонуклеозидтрифосфаты могли спариваться с родительской матричной цепью. Однако в обычных условиях двойная спираль ДНК весьма стабильна; спаренные основания соединены столь прочно, что для разделения двух цепей ДНК в пробирке требуются температуры, приближающиеся к точке кипения воды (90°С). По этой причине большинство ДНК-полимераз может копировать лишь ту молекулу ДНК, у которой матричная цепь уже отделилась от другой цепи. Для того чтобы двойная спираль ДНК раскрылась и соответствующая матричная цепь стала доступной для ДНК-полимеразы, необходимы особые белки. Они бывают двух типов.
ДНК-геликазы были впервые выделены как белки, которые, присоединяясь к одиночной цепи ДНК, катализируют гидролиз АТР. Как уже отмечалось в гл. 3, гидролиз АТР может циклическим образом изменять форму молекулы белка, вследствие чего белок будет производить механическую работу (см. разд. 3.4.11). Именно этот принцип лежит в основе быстрого перемещения ДНК-геликаз по одиночной цепи ДНК. Встречая на своем пути участок двойной спирали, эти ферменты продолжают двигаться вдоль своей цепи и тем самым расплетают двойную спираль (рис. 5-44). Расплетание ДНК-спирали в области репликационной вилки, вероятно, осуществляется двумя совместно действующими ДНК- геликазами, одна из которых перемещается по ведущей цепи, а другая - по отстающей. Ясно, что две эти геликазы должны двигаться вдоль одиночных цепей ДНК в противоположных направлениях, т. е. это должны быть разные ферменты. Действительно, оба указанных типа ДНК- геликаз удалось обнаружить. При этом исследования на бактериях показали, что главную роль играет ДНК-геликаза отстающей цепи. Причины этого мы обсудим ниже.
Белки, дестабилизирующие спираль (их называют также белками, связывающими одноцепочечную ДНК или SSB-белками), связываются с одиночными цепями ДНК, не закрывая оснований, т. е. оставляя их доступными для спаривания. Сами они не способны расплетать длинные молекулы ДНК, но, присоединяясь к одиночным цепям ДНК, они тем самым способствуют любому процессу расплетания спирали; они, например, помогают ДНК-геликазе расплетать двойную спираль в репликационной вилке. На матрице отстающей цепи SSB-белки кооперативным образом связываются с одноцепочечными участками ДНК и предотвращают здесь образование «шпилек», небольших двухспиральных структур, которые могли бы помешать синтезу ДНК, осуществляемому ДНК-полимеразой (рис. 5-45).
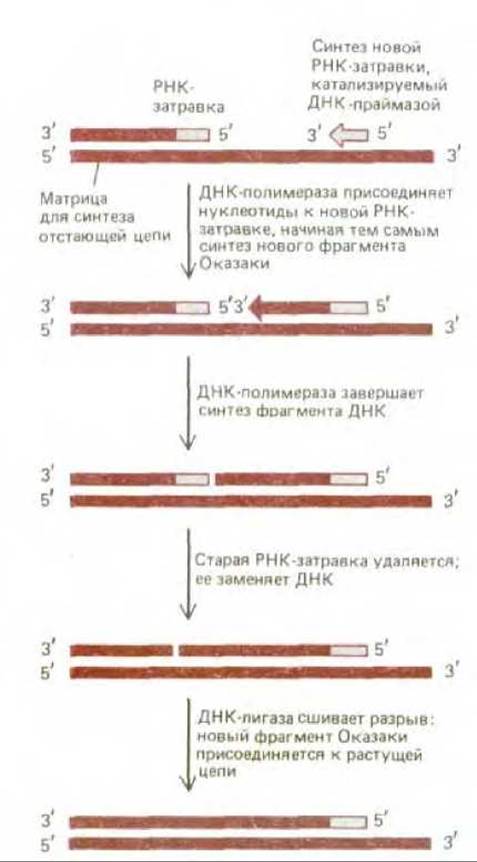
Рис. 5-43. Отдельные этапы синтеза каждого из фрагментов отстающей цепи ДНК. У эукариот РНК-затравки синтезируются в отстающей цепи с интервалами приблизительно в 200 нуклеотидов и каждая из них состоит из 10 нуклеотидов.
5.3.7. Белки в репликационной вилке действуют кооперативно, образуя «репликационную машину» [30]
До сих пор мы говорили о репликации ДНК так, как если бы она осуществлялась смесью репликационных белков, действующих независимо друг от друга. Между тем в действительности большая часть этих белков объединена в крупный мультиферментный комплекс, быстро движущийся вдоль ДНК. Этот комплекс - нечто вроде крошечной «швейной машины»: «деталями» его служат отдельные белки, а источником энергии - реакция гидролиза нуклеозидтрифосфата. Комплекс изучен достаточно хорошо только у бактерий Е. coli и у некоторых вирусов, но есть все основания считать, что очень похожий механизм действует и у эукариот (см. разд. 9.3.3).
Схема на рис. 5-46, где подробно изображена репликационная вилка, позволяет судить о том, как работают отдельные части такой «репликационной машины». В области вилки действуют две идентичные ДНК-полимеразы - на ведущей и на отстающей цепи. Спираль ДНК расплетается в результате совместного действия ДНК-полимеразы, работающей на ведущей цепи, и ДНК-геликазы, движущейся вдоль отстающей цепи; этому процессу способствуют кооперативно связывающиеся молекулы дестабилизирующегося белка. В то время как на ведущей цепи ДНК- полимераза работает непрерывно, на отстающей цепи фермент через определенные интервалы прерывает и вновь возобновляет свою работу, используя для полимеризации короткие РНК-затравки, синтезируемые ДНК-праймазой.
Эффективность репликации сильно возрастает вследствие тесного объединения всех этих белковых компонентов. Молекула праймазы непосредственно сцеплена с ДНК-геликазой, образуя вместе с нею на отстающей цепи структуру, называемую праймосомой, которая движется с репликационной вилкой и по ходу своего движения синтезирует РНК-затравки. Молекула ДНК-полимеразы, работающая на отстающей цепи, также движется совместно с остальными белками, синтезируя ряд новых фрагментов Оказаки; ради этого, как полагают, цепь ДНК, которая служит для нее матрицей, складывается сама на себя, как это показано на рис. 5-47. Репликационные вилки оказываются, таким образом, объединены в одну крупную структуру (с общей массой > 106 дальтон), быстро перемещающуюся вдоль ДНК и обеспечивающую возможность координированного и эффективного синтеза ДНК на обет ветвях вилки.
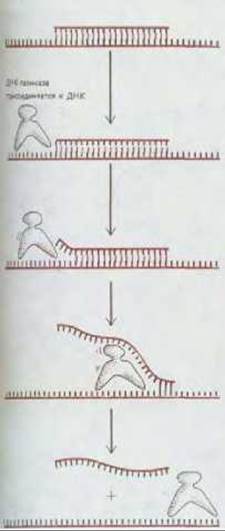
Рис. 5-44. Действие ДНК-геликаз. Небольшой фрагмент ДНК присоединен путем отжига к длинной одноцепочечной ДНК, так что образовался короткий участок двойной спирали. Эта спираль расплетается по мере того, как геликаза движется вдоль одиночной цепи ДНК, катализируя реакцию, для которой требуется наряду с ферментом и АТР. Источником энергии для движения геликазы служит гидролиз АТР (см. рис. 3-63).
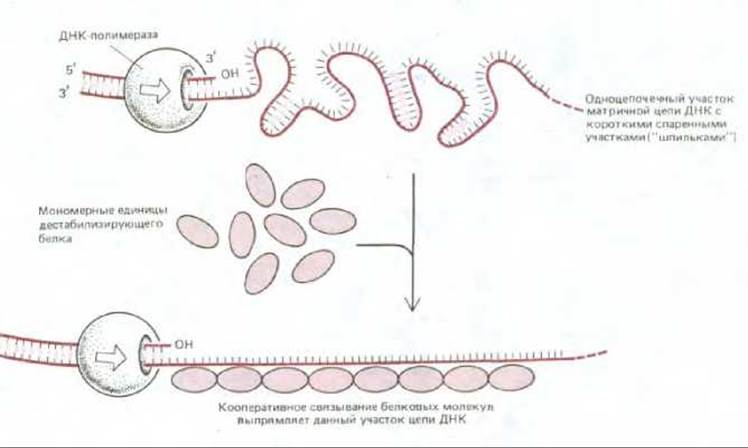
Рис. 5-45. Влияние дестабилизирующих белков на структуру одно-цепочечной ДНК. Поскольку каждая белковая молекула предпочитает связываться с другой, уже связавшейся ранее молекулой (так называемое кооперативное связывание), эти белки образуют длинные кластеры, выпрямляющие матричные пени ДНК и облегчающие процесс полимеризации. Структуры в форме «шпильки», возникающие в свободной одноцепочечной ДНК, образуются путем случайного спаривания оснований в коротких участках, содержащих взаимно комплементарные последовательности нуклеотидов, они напоминают короткие спирали, возникающие во всех молекулах РНК.
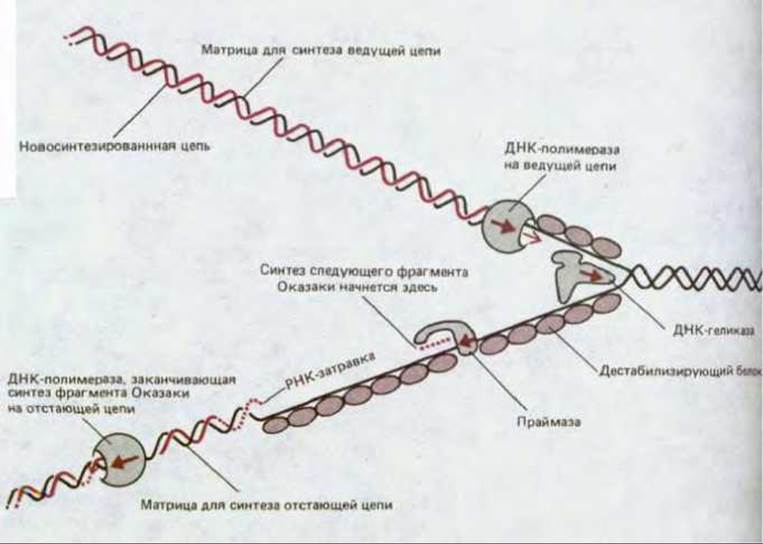
Рис. 5-46. Главные типы белков, действующих в области репликационной вилки (схема показывает их локализацию на ДНК). Комплекс ДНК-праймазы и ДНК-геликазы на отстающей цепи ДНК известен под названием праймосомы.
Позади «репликационной машины» по ходу ее движения остается на отстающей цепи ряд несшитых фрагментов Оказаки, все еще содержащих на своем 5'-конце РНК-затравки, необходимые для инициации на синтеза. Эти РНК-затравки должны быть удалены, а фрагменты сшиты при помощи репарирующих ферментов, работающих позади репликационной вилки (см. рис. 5-43).
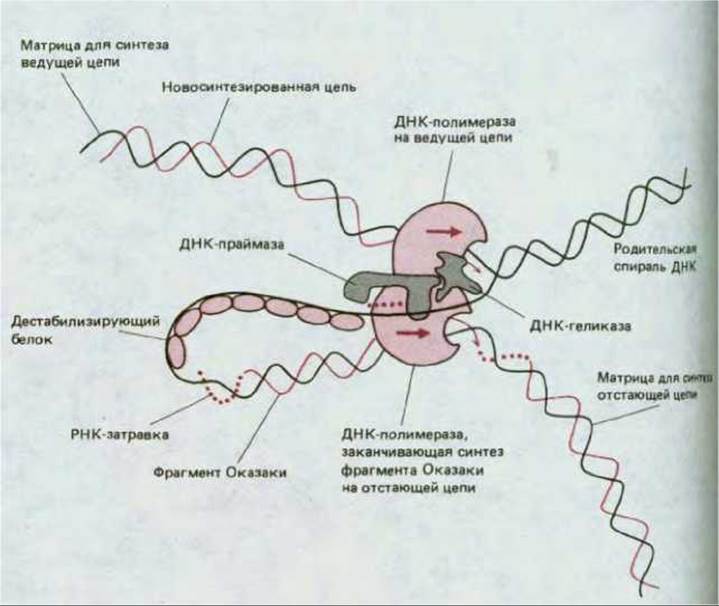
Рис. 5-47. Схема, иллюстрирующая современные представления о расположении репликационных белков в движущейся репликационной вилке. Вместо двумерной структуры, изображенной на рис. 5-46, здесь показано, как ДНК на отстающей цепи складывается, в результате чего возникает комплекс из двух ДНК-полимераз - ведущей и отстающей цепи. Кроме того, благодаря складыванию 3'-конец каждого завершенного фрагмента Оказаки оказывается рядом со стартовым участком следующего такого фрагмента (ср. с рис. 5-46). Находясь в тесном контакте с остальными репликационными белками, молекула ДНК-полимеразы отстающей цепи может непрерывно работать на одной и той же репликационной вилке, отделяясь от готового фрагмента ДНК, она переходит к ближайшей новой РНК-затравке, чтобы начать синтез следующего фрагмента. Обратите внимание, что на этой схеме одна из дочерних спиралей ДНК направлена вправо и вниз, а вторая влево и вверх.
5.3.8. Ошибки при репликации ДНК в бактериальных клетках устраняются особой корректирующей системой, распознающей неправильное спаривание оснований
У таких бактерий, как Е. coli, деление происходит каждые 30 мин, поэтому у них сравнительно легко выявить в большой популяции клеток редкие экземпляры с измененными признаками. Выделен, например, класс мутантов, характеризующихся резким повышением частоты спонтанных мутаций, что связано с присутствием в его клетках специфических генов-мутаторов. Известен ген-мутатор, кодирующий дефектную форму 3' → 5'-корректирующей экзонуклеазы, представляющей собой субъединицу ДНК-полимеразы (см. разд. 5.3.3). Если дефект затрагивает этот белок, то ДНК-полимераза утрачивает способность эффективно осуществлять коррекцию и в ДНК накапливается много ошибок, которые при нормальной репликации были бы устранены.
Изучение тех мутантов Е. coli, у которых имеются гены-мутаторы, выявило еще одну систему, в норме устраняющую ошибки репликации, не улавливаемые корректирующей экзонуклеазой. Эта система коррекции неправильного спаривания (mismatch proofreading sistem), называемая также системой исправления ошибок спаривания (mismatch repair system), отличается от ранее рассмотренных систем репарации ДНК тем, что она не зависит от присутствия в ДНК аномальных нуклеотидов, которые должны быть распознаны и удалены («вырезаны»). Она выявляет деформации на внешней стороне спирали, вызванные плохой пригонкой обычных, но некомплементарных оснований. Если бы эта корректирующая система просто распознавала ошибки спаривания в реплицировавшейся ДНК и удаляла без выбора один из двух неправильно спарившихся нуклеотидов, то в половине случаев она бы сама совершала ошибку, «исправляя» не новосинтезированную, а матричную цепь, так что в среднем частота ошибок оставалась бы прежней. Для эффективной коррекции система должна уметь различать неправильно спаривающиеся нуклеотиды и избирательно удалять такие нуклеотиды только из новой цепи (т.е. устранять именно ошибки репликации).
В клетках Е. coli процесс распознавания связан с метилированием определенных остатков аденина в ДНК. Метальные группы присоединяются ко всем остаткам А в последовательности GATC, но лишь спустя некоторое время после того, как А включится в новосинтезированную цепь ДНК. Новые цепи отличаются от старых тем, что только в них сразу же за репликационной вилкой могут находиться еще не метилированные последовательности GATC. Коррекция неправильного спаривания осуществляется крупным мультиферментным комплексом, сканирующим каждую из двух цепей двойной спирали ДНК. Этот комплекс удаляет только неправильно присоединенные нуклеотиды, но делает это лишь после того, как на той же цепи обнаружится и неметилированная последовательность GATC. Поэтому нуклеотиды удаляются только из новой цепи, т. е. устраняются ошибки репликации (рис. 5-48).
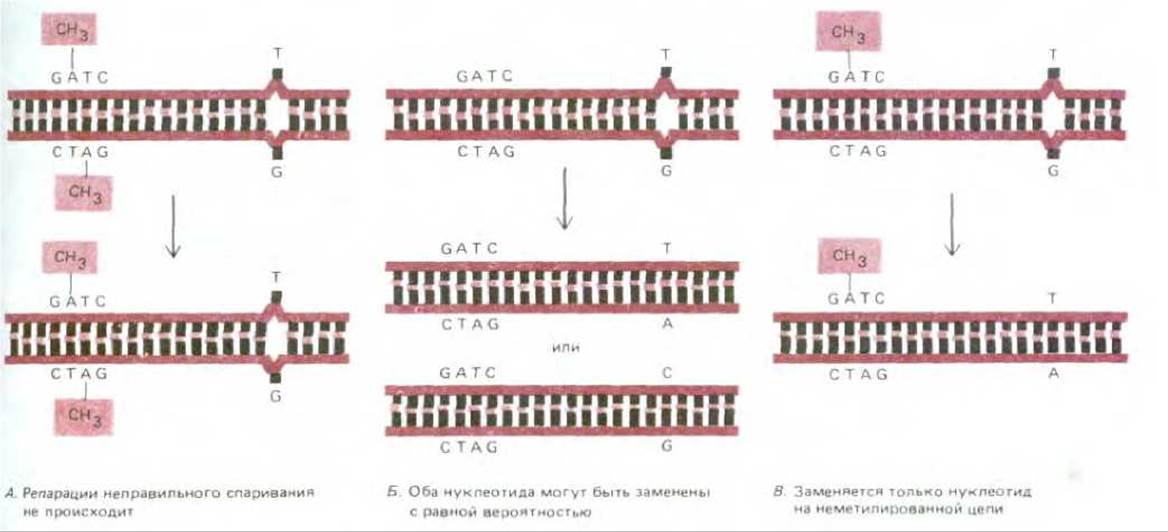
Рис. 5-48. Схема эксперимента, иллюстрирующего работу системы коррекции неправильного спаривания, устраняющей у бактерий ошибки репликации ДНК. Особый белковый комплекс удаляет неспаренные нуклеотиды из вновь синтезируемой цепи ДНК позади репликационной вилки, этот репарирующий комплекс узнает новую цепь ДНК по обнаруживаемым в ней неметилированным последовательностям GATC. На схеме представлены три молекулы ДНК с одной и той же «неправильной» парой нуклеотидов, но при этом в одной молекуле (А) метилированные последовательности GATC встречаются в обеих цепях, в другой молекуле (Б) таких метилированных последовательностей нет совсем, а в третьей (В) они присутствуют только в одной из цепей. Если воздействовать на эти молекулы ДНК клеточным экстрактом, содержащим корректирующий комплекс, то мы получим представленный здесь результат. Молекула ДНК в правой части рисунка воспроизводит картину, обнаруживаемую непосредственно за репликационной вилкой: нижняя цепь соответствует новой цепи, где метилирование еще не произошло.
В эукариотических клетках не удалось пока выявить ни одного из этих двух механизмов коррекции, обнаруженных у бактерий. Однако степень точности репликации у млекопитающих и у Е. coli приблизительно одинакова, и потому можно думать, что оба описанных типа коррекции существуют и у эукариот. Следует, впрочем, отметить, что в ДНК млекопитающих нет метилированных остатков А, поэтому механизм, который используется системой репарации ошибок спаривания для узнавания новосинтезированной цепи, должен быть в данном случае иным.
5-27
5.3.9. Репликационные вилки возникают в точках начала репликации [32]
И у бактерий, и у млекопитающих образование репликационных вилок начинается с возникновения особой структуры, называемой репликационным глазком (replication bubble). Это небольшой участок, в котором две цепи родительской спирали ДНК отделились одна от другой и были использованы в качестве матриц для синтеза ДНК (рис. 5-49). Для бактерий и некоторых вирусов, размножающихся в эукариотических клетках, удалось показать, что репликационный глазок образуется в тех местах молекулы ДНК, где находятся специфические нуклеотидные последовательности, получившие название точек начала репликации. Эти последовательности состоят приблизительно из 300 нуклеотидов. Предполагают, что аналогичные точки начала репликации существуют и в эукариотических хромосомах, однако надежных доказательств этого пока нет (см. разд. 9.3.2).
Процесс возникновения репликационных вилок удалось в некоторых случаях воспроизвести in vitro. Эти опыты показали, что у бактерий и бактериофагов инициация репликационных вилок начинается так, как это представлено на рис. 5-50. Множество копий инициаторного белка связываются с особыми участками в точке начала репликации, образуя крупный белковый комплекс. Этот комплекс присоединяет затем ДНК-геликазу и помещает ее на свободную одиночную цепь ДНК в прилегающем участке спирали. Присоединяется также ДНК-праймаза, т. е. образуется праймосома, которая, двигаясь от точки начала репликации, синтезирует РНК-затравку, что дает возможность начать синтез первой цепи ДНК. Остальные белки быстро объединяются после этого в два репликационных белковых комплекса, которые теперь движутся от точки начала репликации в противоположных направлениях (см. рис. 5-49); они продолжают синтезировать ДНК до тех пор, пока обе вилки не пройдут путь по матрице до самого конца.
Некоторые дополнительные данные, касающиеся инициации репликационных вилок в хромосомах эукариот, мы обсудим в гл. 9, там, где речь пойдет о клеточном ядре.
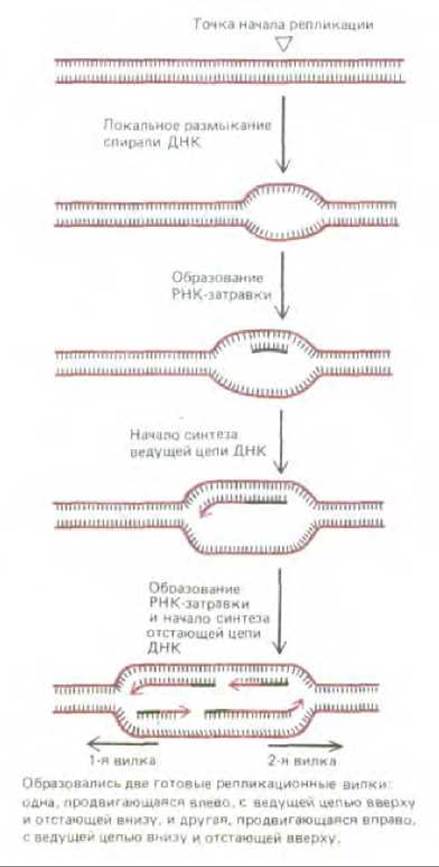
Рис. 5-49. Гипотетический механизм образования репликационных вилок в точках начала репликации (см. также рис. 5-50).
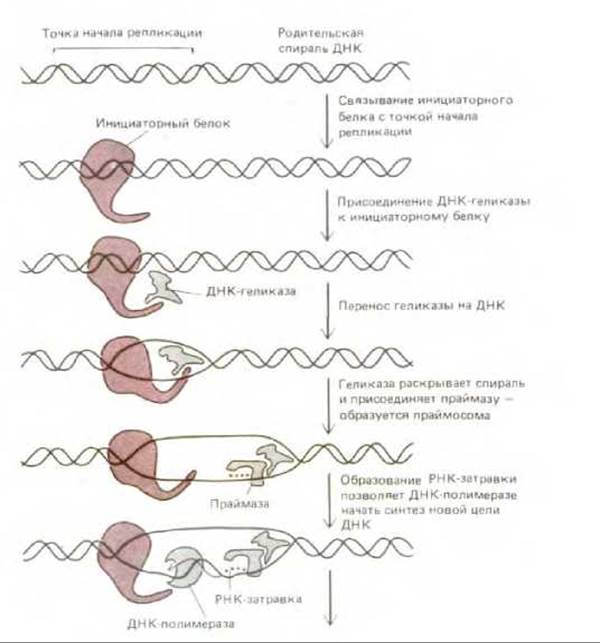
Рис. 5-50. Упрощенная схема, иллюстрирующая начальные этапы образования репликационных вилок в точках начала репликации у Е. coli и бактериофага X. Для обнаружения данного механизма потребовались опыты in vitro с использованием смеси высокоочищенных белков.
Последующие этапы приводят (пока не ясным путем) к инициации еще трех цепей ДНК (рис. 5-49). У Е. coli в репликации ДНК роль инициаторного белка играет белок dnaA; а праймосома состоит из белков dnaB (ДНК-геликаза) и dnaG (ДНК-праймаза).
5.3.10. ДНК-топоизомеразы предотвращают спутывание ДНК во время репликации [33]
Изображая спираль ДНК так, как мы это делали до сих пор, т.е. неправильно, в виде плоской «лестницы», мы игнорировали «проблему закручивания» (winding problem). Между тем на каждые 10 пар оснований, образующихся в репликационной вилке, родительская двойная спираль должна совершить один полный оборот вокруг своей оси. Следовательно, для того чтобы репликационная вилка могла продвигаться вперед, вся хромосома впереди нее должна быстро вращаться (рис. 5-51), что для длинных хромосом потребовало бы большой затраты энергии. При репликации ДНК эта проблема решается иначе: путем образования в спирали своего рода «шарнира», особого класса белков, называемых ДНК- топоизомеразами.
ДНК-топоизомераза представляет собой нечто вроде «обратимой нуклеазы». Сначала она разрывает цепь ДНК, а затем ковалентно присоединяется к разорванному концу. Ковалентная связь белок — ДНК обладает довольно значительной энергией, потому что в ней сохраняется энергия разорванной фосфодиэфирной связи. Вследствие этого реакция, приводящая к разрыву цепи, обратима и не требует дополнительной затраты энергии. В этом отношении данный механизм существенно отличается от механизма действия ДНК-лигазы, о котором мы говорили выше (см. рис. 5-35).
Существуют различные типы ДНК-топоизомераз. Топоизомераза типа I разрывает только одну из двух цепей двойной спирали ДНК, что дает возможность двум участкам ДНК по обе стороны от разрыва свободно вращаться относительно друг друга вокруг фосфодиэфирной связи, находящейся напротив разрыва, которая в этом случае выполняет роль упомянутого выше «шарнира» (рис. 5-52). Всякое напряжение в спирали ДНК заставляет ее вращаться в таком направлении, чтобы ослабить это напряжение. Поэтому вращение во время репликации ДНК происходит лишь на коротком отрезке спирали - в той части, которая находится непосредственно перед репликационной вилкой. Аналогичная проблема, возникшая в процессе транскрипции ДНК, решается таким же путем.
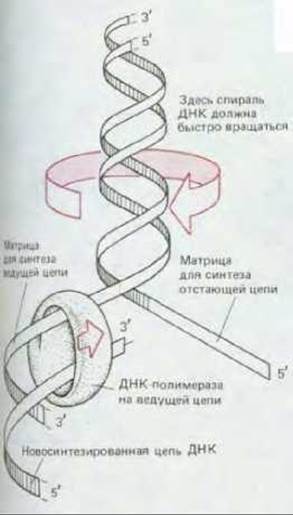
Рис. 5-51. «Проблема кручения», возникающая при репликации ДНК. Для того чтобы репликационная вилка (у бактерий) могла продвигаться вперед со скоростью 500 нуклеотидов в 1 с, родительская спираль ДНК перед вилкой должна вращаться со скоростью 50 об/с.
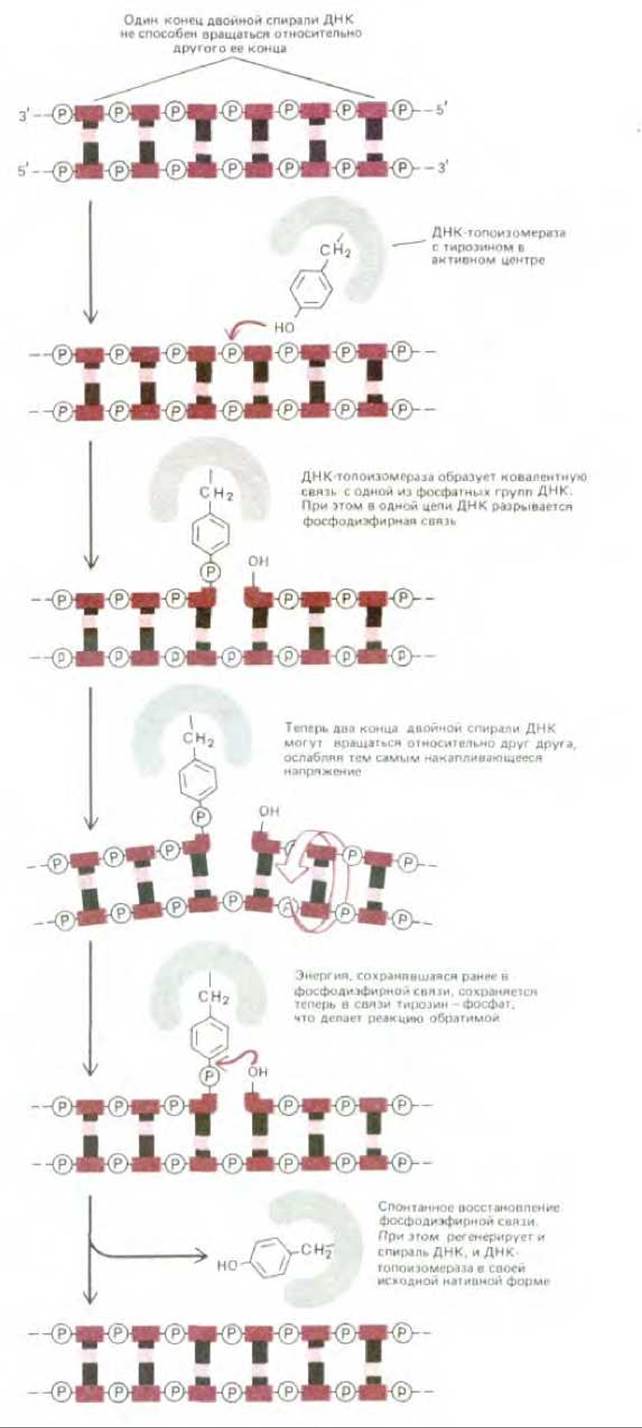
Рис. 5-52. Обратимая реакция, приводящая к появлению разрыва в одной из цепей ДНК. Реакция у эукариот катализируется ДНК-топоизомеразой типа I Ферменты этой группы образуют временную ковалентную связь с ДНК.
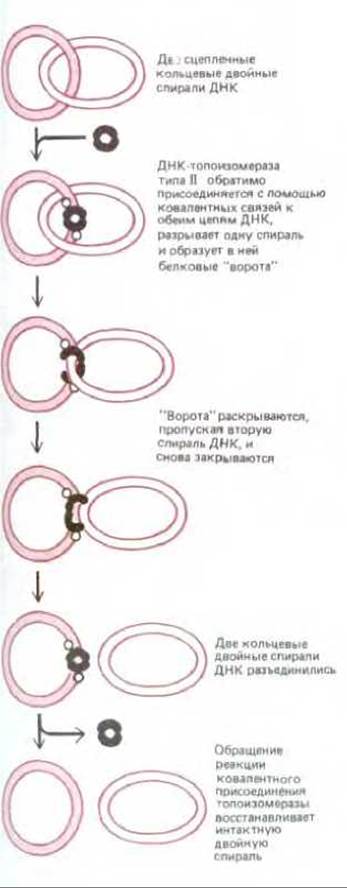
Рис. 5-53. Пример реакции разделения двух сцепленных кольцевых молекул ДНК, катализируемой ДНК-топоизомеразой типа II. Действие этих ферментов (в отличие от реакций, катализируемых ДНК-топоизомеразами типа I) сопряжено с гидролизом АТР и некоторые из них способны сообщать спирали ДНК дополнительное напряжение. ДНК-топоизомеразы типа II обнаруживаются и у прокариот, и у эукариот, по всей вероятности, они участвуют во многих реакциях, имеющих отношение к ДНК.
Топоизомераза типа II ковалентно связывается с обеими цепями двойной спирали ДНК и вносит в нее на время двухцепочечный разрыв. Ферменты этого типа активируются под действием тех участков на хромосомах, где перекрестились спирали. Присоединившись к такому перекресту, топоизомераза: 1) обратимо разрывает одну из двух двойных спиралей, создавая тем самым для другой своего рода «ворота», 2) вынуждает вторую двойную спираль пройти через этот разрыв и 3) сшивает обе разорванные цепи, а затем отделяется от ДНК. Действуя подобным образом, топоизомеразы типа II очень быстро разделяют две сцепленные кольцевые молекулы ДНК (рис. 5-53). Точно так же предотвращают они и спутывание молекул ДНК, которое в противном случае неизбежно создавало бы при репликации серьезную проблему. Известны температурочувствительные мутанты дрожжей, вырабатывающие топоизомеразу II, которая при 37°С инактивируется. Если нагреть эти дрожжевые клетки до такой температуры, то их хромосомы в процессе митоза остаются спутанными и не могут разойтись. Насколько необходим для распутывания хромосом такой «инструмент», как топоизомераза II, поймет каждый, кто хоть раз пытался распутать безнадежно запутавшуюся леску, не имея под рукой ножниц.
5-28
5.3.11. Репликация ДНК у эукариот и прокариот в основных чертах сходна [24]
Почти все, что мы знаем о репликации ДНК, удалось выяснить в опытах с очищенными мультиферментными системами бактерий и бактериофагов, способными осуществлять репликацию ДНК in vitro. Получение таких систем в 1970-х годах заметно облегчилось после того, как удалось выделить мутанты по целому ряду различных генов, ответственных за репликацию, которые можно было использовать для идентификации и очистки соответствующих белков (рис. 5-54).
У эукариот энзимология репликации ДНК пока еще детально не изучена, главным образом потому, что получать здесь необходимые мутантные формы гораздо труднее. Однако схема репликации у прокариот и эукариот в основных чертах, включая геометрию репликационной вилки и потребность в РНК-затравке, по-видимому, одинакова. Главное различие заключается в том, что у эукариот ДНК реплицируется не как таковая, а в виде хроматина, в котором она прочно связана с белками, принадлежащими к классу гистонов. В гл. 8 мы узнаем, что гистоны образуют комплексы в форме дисков, вокруг которых обвивается эукариотическая ДНК, в результате чего возникают регулярно повторяющиеся структуры, называемые нуклеосомами. Нуклеосомы располагаются вдоль молекулы ДНК с интервалами 200 пар оснований. Быть может, именно этим объясняется тот факт, что новые фрагменты отстающей цепи ДНК закладываются у эукариот с интервалами в 10 раз более короткими (от 100 до 200 нуклеотидов), чем у бактерий (от 1000 до 2000 нуклеотидов). Кроме того, если нуклеотиды служат барьерами, на время останавливающими продвижение ДНК-полимеразы, присутствие хроматина (а не голой ДНК) может, вероятно, объяснить и то, что репликационные вилки движутся у эукариот приблизительно в 10 раз медленнее, чем у бактерий.
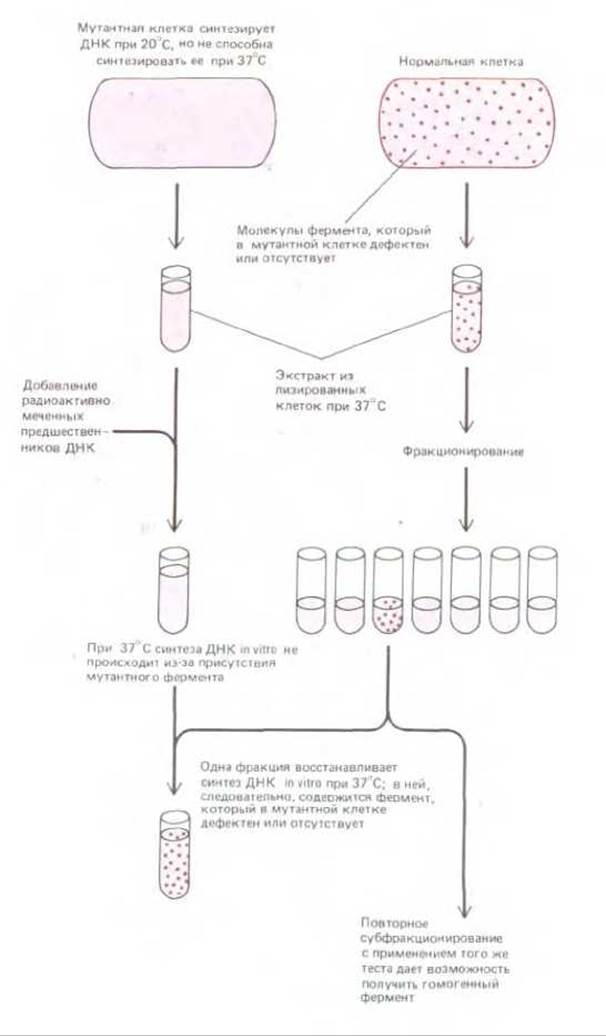
Рис. 5-54. Получение у бактерий и бактериофагов мутантов с различными нарушениями репликации ДНК открыло возможности для выявления и очистки ферментов, выполняющих какую-либо еще не известную функцию, необходимую для репликации ДНК у прокариот. Использованные здесь температурочувствительные мутанты принадлежат к так называемым условным мутантам, обычно их фермент нормально функционирует при низкой температуре и не работает при высокой. У «безусловных» мутантов с нарушениями репликации синтез ДНК не идет ни при низкой, ни при высокой температуре, и потому эти мутанты обречены на гибель. В модифицированной форме такие «тесты на комплементацию in vitro» полезны также при биохимическом изучении многих других процессов.
Заключение
Самокорректирующая ДНК-полимераза катализирует полимеризацию нуклеотидов на обеих цепях спирали ДНК в направлении 5' → 3', копируя матрицу с высокой степенью точности. Поскольку две цепи двойной спирали ДНК аптипараллелъны, в направлении 5' → 3' может непрерывно синтезироваться лишь одна из двух цепей (ее называют ведущей). Другая, отстающая цепь синтезируется в виде коротких фрагментов по принципу «шитья назад иголкой». Самокорректирующая ДНК-полимераза не способна начинать синтез новой цепи. Поэтому для закладки фрагментов отстающей цепи ДНК используются короткие молекулы РНК-затравки, которые позже удаляются - их заменяет ДНК.
Процесс репликации ДНК требует совместного действия многих белков. В нем участвуют: 1) ДНК-полимераза и ДНК-праймаза, катализирующие полимеризацию нуклеозидтрифосфатов; 2) ДНК-геликазы и дестабилизирующие белки, помогающие расплести спираль ДНК, которую предстоит копировать; 3) ДНК-лигаза и фермент, разрушающий молекулы РНК-затравки; они нужны для сшивания прерывисто синтезируемых фрагментов отстающей цепи ДНК; 4) ДНК-топоизомеразы, помогающие решить проблемы кручения и спутывания спирали ДНК; 5) инициаторные белки, присоединившиеся к специфическим последовательностям ДНК в точке начала репликации и способствующие образованию здесь новой репликационной вилки. В точке начала репликации к ДНК-матрице сначала присоединяется белковый комплекс, состоящий из ДНК- геликазы и ДНК-праймазы (его называют праймосомой); затем к этому комплексу добавляются другие белки и возникает мультиферментный комплекс — «репликационная машина», которая и осуществляет синтез ДНК.